文献解读:纵向保守性微生物互作网络
题目:A sparse covarying unit that describes healthy and impaired human gut microbiota development
通讯作者:Jeffrey I.Gordon(美国科学院院士)
杂志:Science
发表年月:2019.7
简介:19年7月发表在Science上的文章,Jeffrey I.Gordon团队同期还有另一篇文章也发表在Science上。两篇文章结合起来阅读是非常好的微生物组学学习材料(无论是科研思路,数据分析还是写作上)。最重要的一点,学习大牛是怎么把基础研究成果转化到临床上的。
名词解释
SAM/MAM:severe/moderate acute malnutrition,严重/中度急性营养不良。
MDCF:microbiota-directed complementary foods ,靶向微生物的补充食品
RUSF:ready-to-use supplementary food,即食补充食品。
Ecogroup:生态群,由作者定义的保守分类单元。
研究目的
本篇文章中作者提出了一种新的构建网络的方法,为了更好的理解作者的初衷,最好先阅读作者的另一篇同期Science文章(Effects of microbiota-directed foods in gnotobiotic animals and undernourished children,简称paper2),在这篇文章中提到了作者的最终目的:开发针对性调控肠道微生物的治疗食品,用于治疗sam/mam儿童。下面简单介绍一下paper2的背景:
背景1(菌群与年龄的关系):前人的研究利用随机森林回归器,从正常出生儿童队列发现了一些年龄判别菌(特征)。本文作者受此启发,利用测序得到的SAM/MAM微生物组数据发现这些儿童的菌群在RF回归器下预测得到的年龄普遍小于他们的实际年龄,显得更“年轻”。
背景2(发育不良与肠道菌群的因果关系):前人的研究把发育不良儿童和健康儿童的粪便移植到无菌小鼠中,发现移植了前者的小鼠会出现一些生理上的异常。他们同样构建了年龄判别器,发现年龄判别器也是生长判别器。(一个是回归预测年龄,一个是分类判别是健康还是营养不良,后者发现的特征是前者的子集)。研究人员随后把生长判别菌移植到异常小鼠肠道中,发现能够改善表型。证明了菌群与发育存在因果关系。
因此,一个自然的想法是:既然存在因果关系,那么是否可以通过调节肠道菌群把SAM/MAM儿童的菌群结构拉回正常儿童应有的年龄?
背景3:根据世界卫生组织的建议,母乳喂养在减少儿童营养不良方面起着重要作用。他们建议在出生头6个月进行纯母乳喂养,6-24个月(称为断奶期)继续母乳喂养并引入补充食品。然而,尚无人针对调控肠道菌群的发育过程去开发补充食品。
因此,作者的最终目的就是想通过筛选得到特定的食品成分,这些成分能够选择性的增加在SAM.MAM儿童中缺失的年龄-生长判别菌的丰度,使SAM/MAM儿童的肠道菌群结构向正常儿童菌群结构靠近。又考虑到前人研究到的因果关系,如果菌群回到正常年龄,不就可以改善营养不良了吗?
有了这个想法就有了第二篇文章,即怎么一步步从小鼠,猪动物实验中筛选得到食品再到临床试验的过程,这里暂不详细介绍。
值得一提的是,怎么衡量开发出来的补充食品对肠道微生物的修复程度?这其实是一个另一个科学问题,也就有了paper1。一开始,根据前人研究以及作者的数据发现,SAM/MAM儿童肠菌年龄偏低,一个自然的想法是想利用随机森林模型预测得到的肠菌年龄来作为一个修复衡量指标。然而,实践中他们发现,因为MAM儿童的肠菌年龄基线较大(意思是与正常儿童离得很近,导致预测标准误差必须很小才行否则预测得到的没有意义),所以这个方法就行不通。于是他们转而利用构建跨时间尺度的保守性网络的方法,去解答这个问题,这就有了paper1, 下面介绍paper1。
研究介绍
前面曾提到作者会写出这篇文章的一个最主要的目的是开发一种能够表征接受了治疗食物干预后的MAM/SAM肠菌修复程度的方法(即保守性网络),这是初心。但在这篇文章的写作过程中,背景介绍则扩展成了围绕保守性网络的优点展开。
背景(针对保守性网络方法做的背景介绍):作者把微生物群看作一个由不同零件组成的生态系统。因此,很自然的,有人把菌的丰度作为一个指标,用于衡量系统配置与菌群特征的关系。然而,以往的方法往往忽略了菌之间的相互关系。一个关键问题是如何发现具有生物学作用的交互,这样有助于我们减少用于刻画微生物群的特征数量。(这句话的潜台词是说,如果我们能够构建一个稀疏的但又能高度描述整个系统的网络,那么我们就能以最少的特征数量去刻画整个系统,而不是把系统的全部要素都囊括进来。)
当前的方法
1.传统的共现分析:描述交互作用的能力有限
2.SparCC/SPIEC-EASI(两种近年比较流行的方法)
SparCC原理介绍
SparCC:Sparse Correlations for Compositional data。
基本假设是OTU互作网络是稀疏的(即大多数节点都不相关)。
最简单网络构建方法是直接基于丰度信息计算相关系数(皮尔森,斯皮尔曼),然后根据相关系数的大小设定一个阈值,大于该阈值的就赋予一条边。
SparCC并不是直接根据样本的丰度去计算相关系数,它基于两个otu丰度的log比值的方差去推断相关系数,结果要比普通的方法更鲁棒一些。
为什么要基于log比值?因为我们的数据是相对定量的数据,而相对定量意味着是百分比,需要先进行归一化。如果直接基于丰度去推断相关性,很容易受到归一化的影响。(比如,利用丰度计算时还是正相关,而归一化以后变成了负相关),但是log比值不会被归一化影响。
局限:为横断面研究设计,无法揭示纵向研究(时间序列)的保守性质。
解决思路
借用其他领域的方法:协方差耦合矩阵分解(PCA)。在经济物理学/蛋白质进化领域,该方法能够发现共同波动的经济成分,功能相关的氨基酸协作网络。基本假设:在一个复杂的动态系统中,保守的共变体能够提供有价值的信息。
Part I:识别ECO-GROUP
1.孟加拉国健康儿童队列情况
36个成员,其人体测量得分始终保持健康。
出生后前5年,每月采样一次,一共有1961个粪便样本
此外,还纳入了19个来自Mirpur的儿童,一共收集到2455 个粪便样本。
对16s rRNA基因的V4区进行了扩增子自测序,根据大于97%的同一性进行聚类。
最终,得到了至少在2个样品中丰度大于0.1% **的OTU118个,后续的分析基本都是基于这118个otu。**
2.对肠道的发育过程进行的首要描述
因为想看的是菌群发育,年龄的关系,因此运用unifrac距离对1-60月的儿童样本,以及之前研究的49个23-49岁样本(也是住在Mirpur)一起拿来上看了下样本间的进化相似性。
unifrac:一种beta-多样性的衡量指标,测量两个样本的群落的细菌进化树所共享的分支长度,从而用于刻画样本间的相似性。
![1563765169010]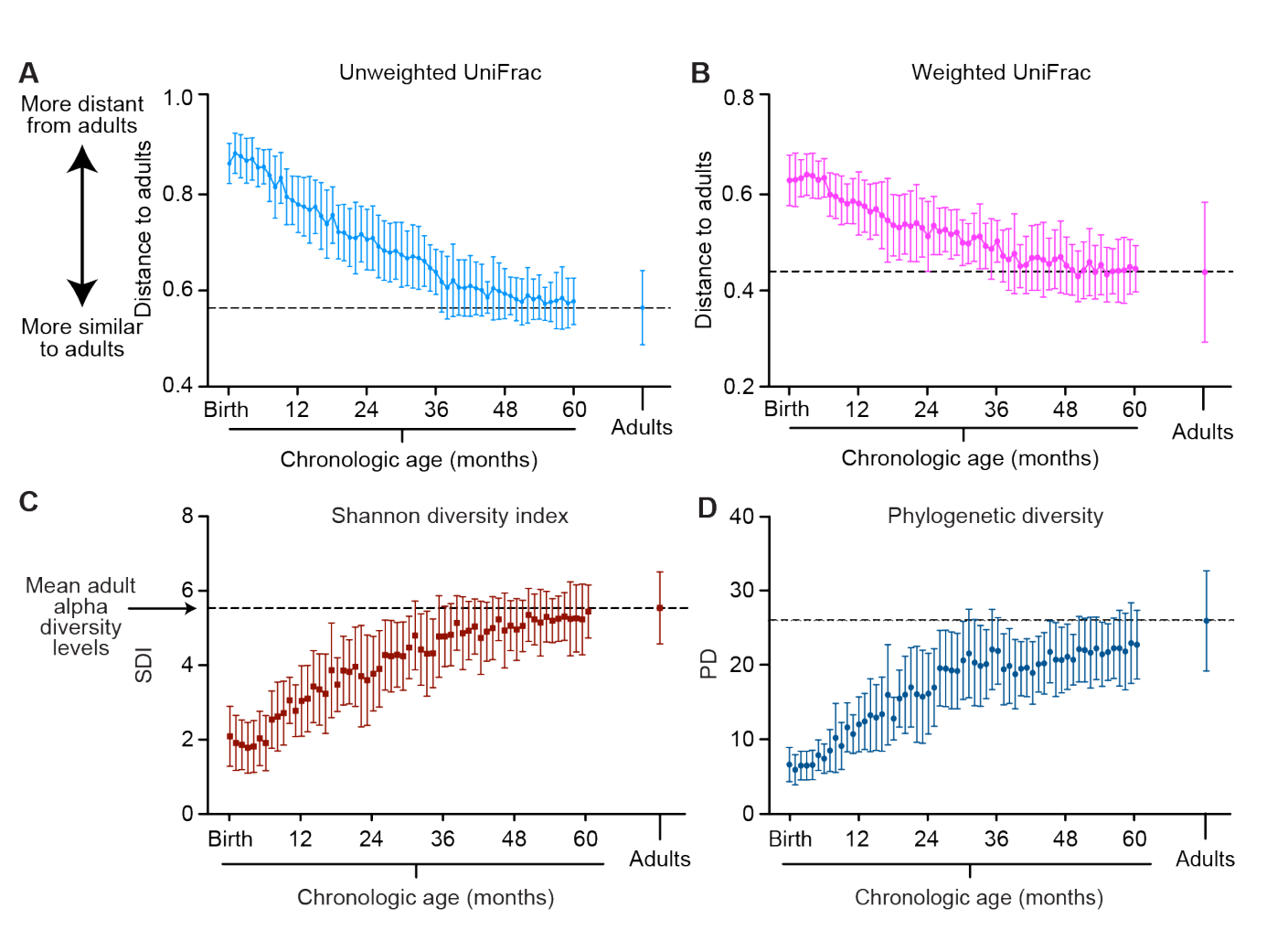
A/B:计算了1-60个月的健康儿童与成人样本的unifrac距离。成人bar是作为参考对照,计算了成人样本间的相似距离。(可以看到,儿童越大,距离越近,从36个月起,离“儿童-成人”的相似性与“成人-成人”的相似性到达了同一个水平;之前也有横断面研究的文章发现这个时间点的儿童菌群配置与成人相似。)
C/D:两种alpha-diversity指标揭露了健康儿童菌群的多样性。
3.随机森林构建年龄判别器
为了进一步探究菌群的发育情况,构建RF模型,模型可以给出预测值——肠道”年龄”。
![1563766696277]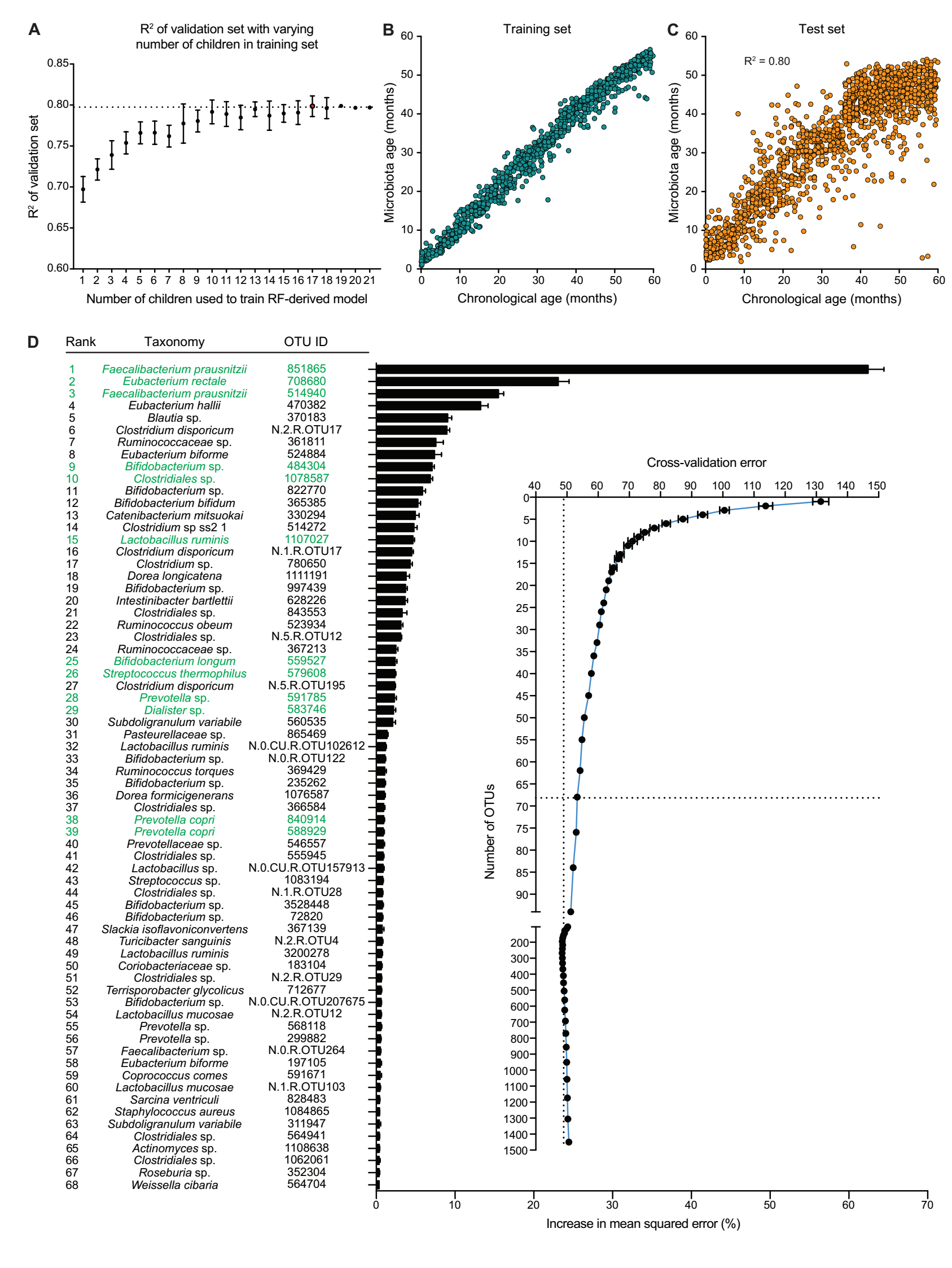
A:随机抽样17个人在测试集上达到了最大的R方,因此选择17个人作为训练集。
B&C:纵轴microbiota-age是判别器预测的年龄,横轴是实际年龄,可以看到在测试集合上,R方=0.8.
D:随机森林的特征选择,特征重要得分。
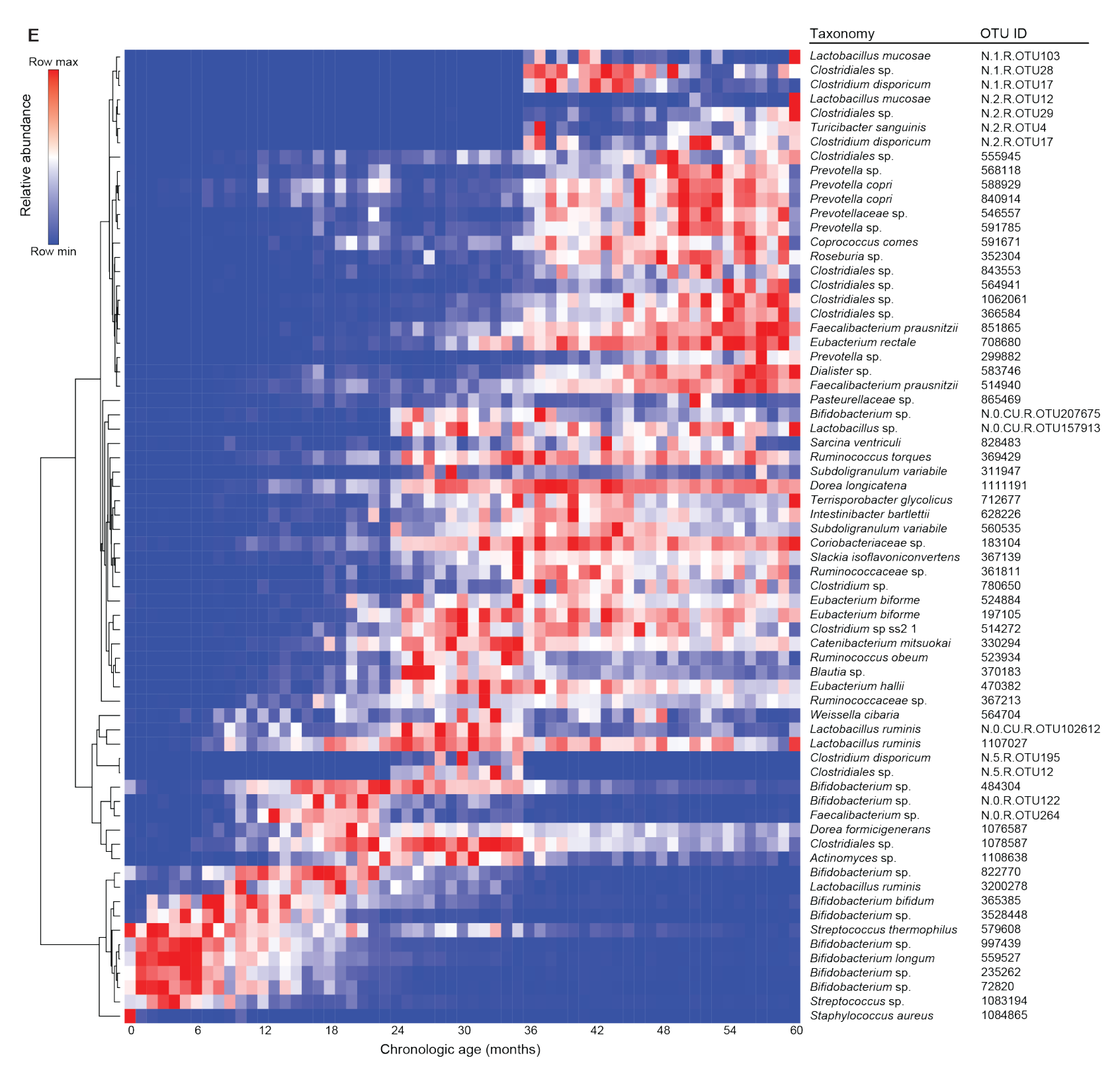
E:年龄判别的taxa的聚类热图
总结:
以上这两个数据分析方法揭示了随年龄变化,肠道微生物的发育过程,并找到了一些marker——称为年龄判别菌。但局限性在于没有表征其相互作关系。解决方案:跟踪主成分谱随时间的变化,这能够描述肠道菌群随时间推移不断演变的过程。
4.迭代PCA(iPCA)
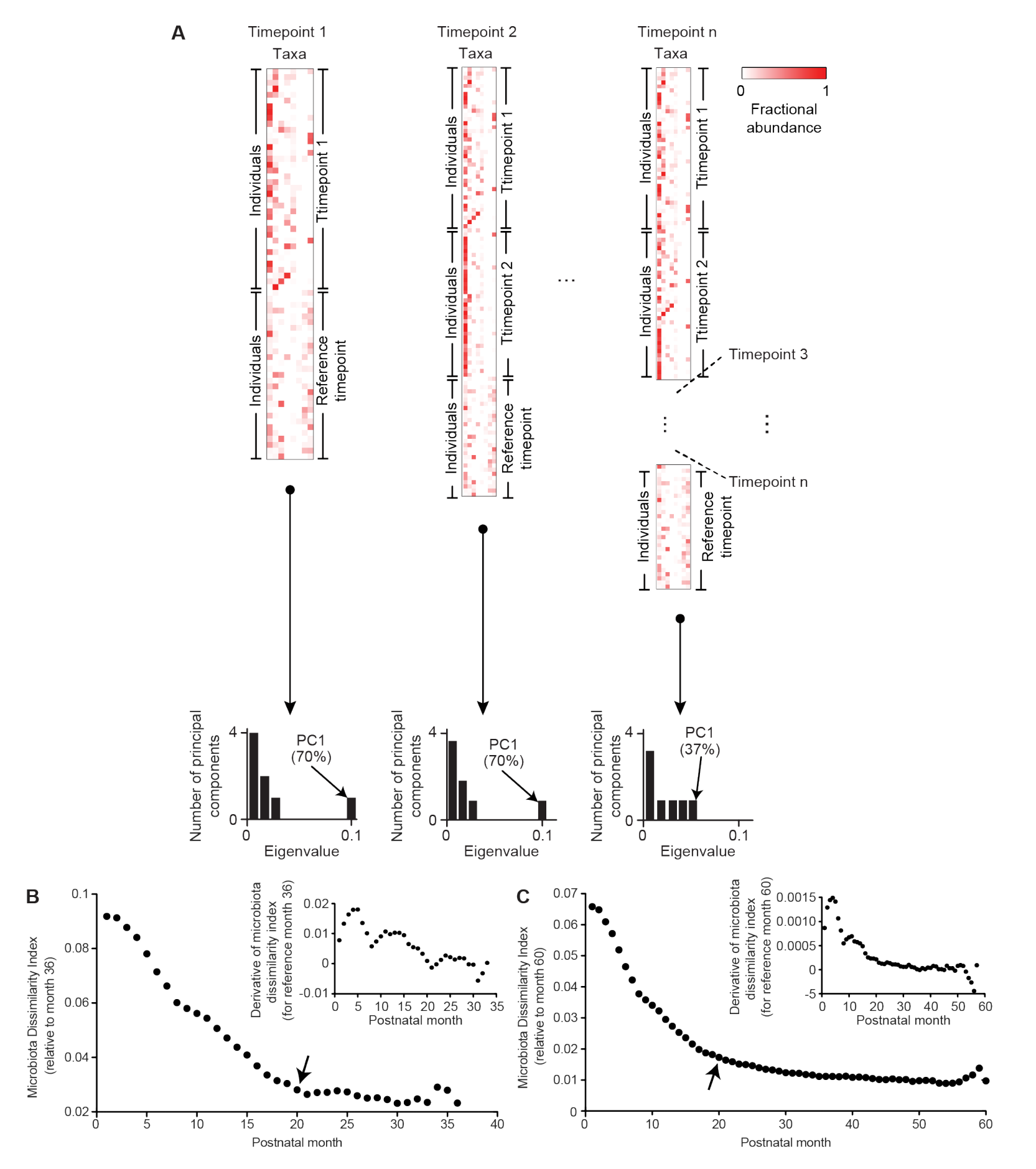
A:每一列是一个otu,一共118列(前面有筛选标准)。两个时间节点的相似性会反映在PC1上(思考:多维数据如何计算相关性,WGCNA是如何计算模块间的相似性的,以及多元统计方法CCA的原理)。
当添加了新的时间节点,却几乎对方差没有增加贡献的时候,可以认为系统趋于稳定。
B:36月作为参考时间点(之前的进化相似性分析发现,36月时与成人的据菌群比较接近)。y轴是作者定义的微生物相异系数,用于表征某个时间点与参考时间点的相似情况,数学上是由iPCA获得的PC1对应的特征值定义。
图B说明了从第20个月起,此后的一系列时间节点带来的结构变异都达到了最小值。
小图表示对时间的导数。(反映变化率,可以看到第20月的变化率最低)
C:与B相似,把第60个月作为参考时间点。得到的结论与B相似。
结论:从图B和C得出结论,从20个月起,到60个月,都没有发生大的结构变异。(这背后的数学原理是PC1代表的方差几乎不再变化,对应的生物学意义在于从20个月起,一些主要的菌群(相对丰度大的)的相对丰度没有再发生什么变化;
下面是一些个人的理解:我们回忆第一张unifrac距离的分析图,会发现作者提到第36个月时的菌群配置才与成人相似,说明从第20个月起,肠道菌群的演化过程主要是一些丰度较小的细枝末节的菌丰度在起着微妙的变化(反映在PC2,3,4…n上),而主要菌群(反映在PC1上)已经基本不再变化。这才导致了20个月时的unifrac距离与成人还有一定差距,而到了36个月的时候,可能这些菌的丰度也趋于稳定)。
因此,这种趋于稳定的特点,允许了作者去设计一个workflow,利用20~60个月的数据计算出一个成熟的微生物群形成过程(参考系是第36个月)中保守的协变体,而不需要对菌的重要程度做出任何先验性假设。
到这一步,重新理一下思路:
作者首先对出生队列,通过与成人菌群对比进行了alpha/beta多样性分析。
观察到第36个月与成人的配置接近。
随后,构建了一个年龄判别器,特征重要性找出了一批相关的菌。但是无法揭示这些菌之间的互作关系。
因此,借助iPCA的方法,发现第20个月起,重要菌的结构已经不再发生变化。
所以,接下来的目的就是通过第20个月起到第60个月的数据,找出那些保守的协变体(再一次强调,其基本假设是这些跨时间尺度的保守协变体起着重要作用)。
5.构建稀疏,共变化网络
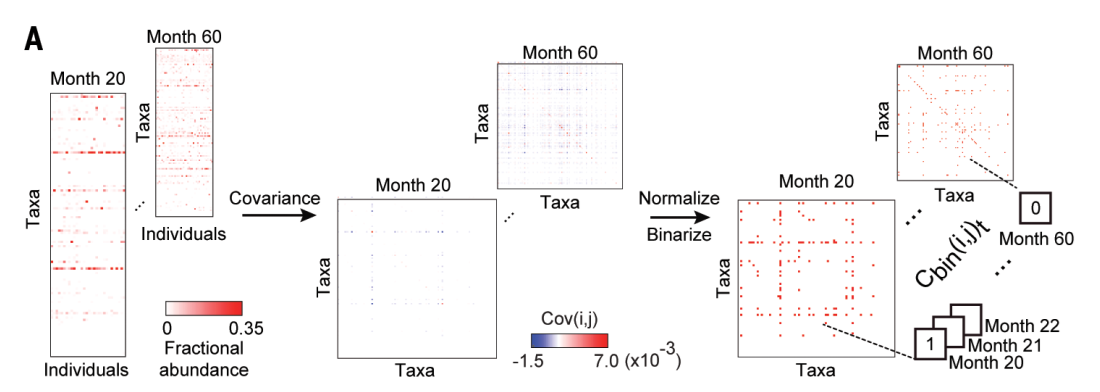
A:每个月的原始矩阵,构建协方差矩阵(反应相关性),进行归一化。并把前10%的,后10%的归为1,其他80%归为0(这种处理表明作者的基本假设是微生物间的互作网络是稀疏的).并把这40个归一化后的矩阵合并一个跨时间尺度的叠加矩阵。如果otu-otu两两之间在叠加矩阵上的相关系数表现的很大,就可以证明这两otu在不同时间尺度都是保守协变的,那这种OTU以及之间的互作关系在动态系统的演化(肠道菌群的发育)上一定是非常重要的。
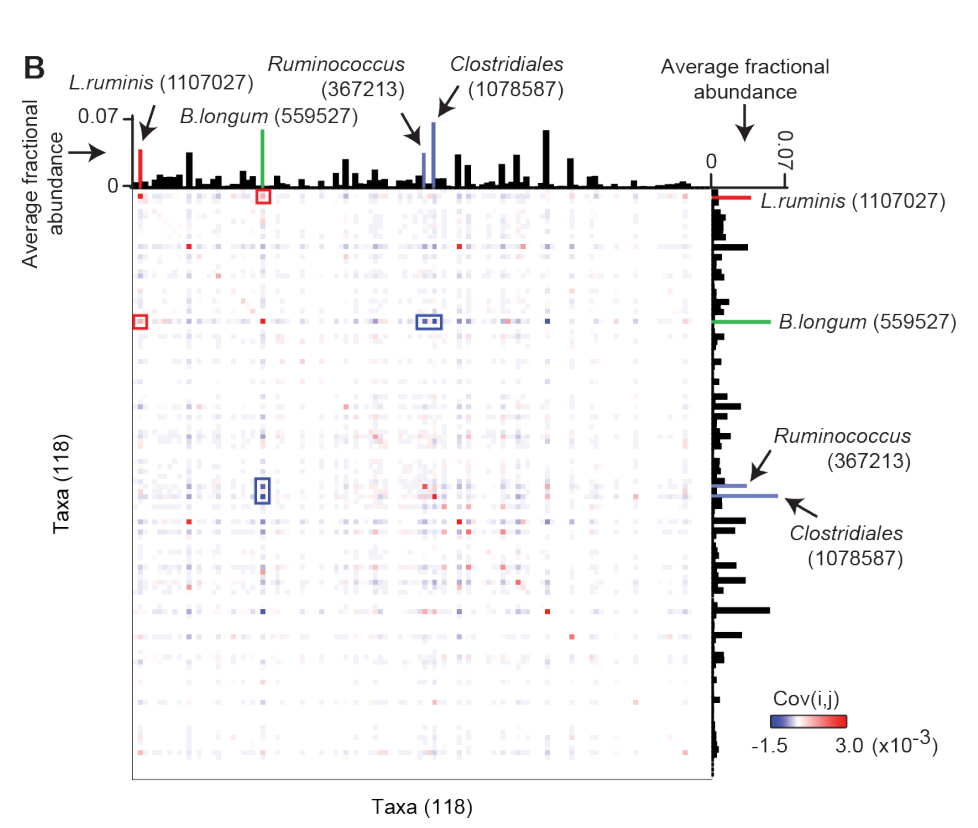
B:叠加矩阵的柱状图+热图。柱状图显示了菌的丰度,热图揭示了跨时间尺度的相关性。比如,B.longum与L.ruminis是正相关(红点),Ruminococcus and Clostridiales 是负相关。此外,从图中也可以看出来大部分的taxa都是独立变异(白色),不具备相关性。
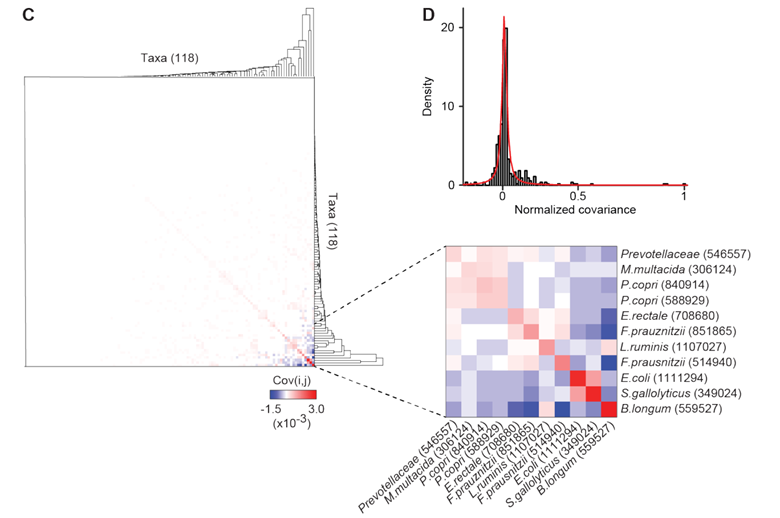
C:对B进一步做层次聚类,揭示了协方差的稀疏性。(大部分都为接近0)
协方差最高的几个如图所示。
D:协方差归一化后的数值分布,再一次看出,在0附近的占了大多数。
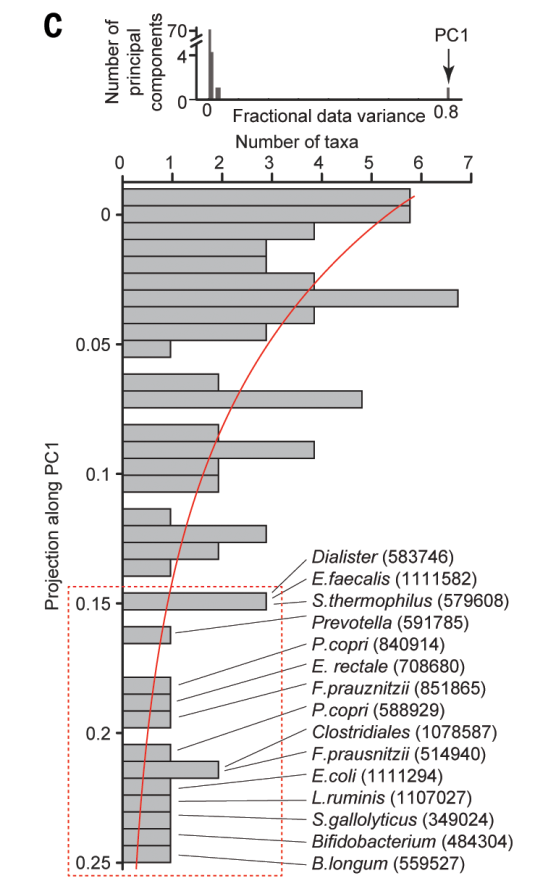
对叠加矩阵进行PCA投影,PC1能够解释80%的变异。(很显然,这80%的变异是由那些显著相关的taxa贡献的,因为中间的80%都被压缩成了0)
取PC1中系数top20%,就得到了15个co-varying taxa,称为ecogroup.这15个taxa全部都在年龄判别器中。
C:纵轴表示投影载荷(可以理解为系数,系数越大,表明对该轴的方差贡献也越大),横轴表示处在该系数下的OTU有几个。
红色方圈的部分是截取了前20%的系数,一共得到了15个OTU。这15个OTU代表了从20~60个月间,在不同时间点上都高度保守具有共变化关系的菌。
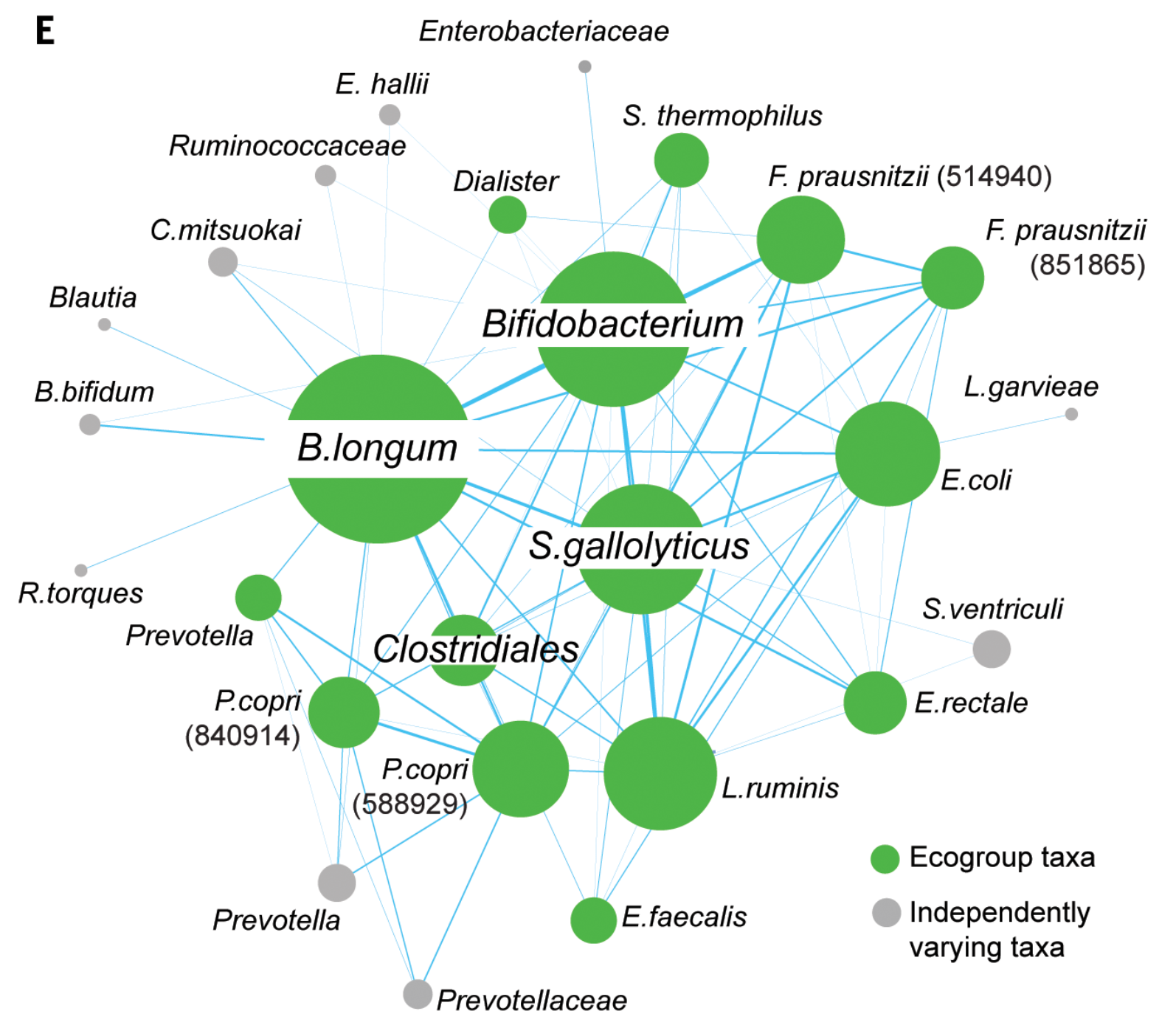
对得到的15个taxa进行网络可视化。
E:边:如果两个节点的cov在叠加矩阵中处于top20%,就连接。点:大小取决于与其相连的边的个数。绿色表示共变化的菌(共15个)。灰色表示非共变化的菌。
到这一步,重新理一下思路:
作者的目的很明确,就是要找出在健康儿童菌群的发育过程中很重要的菌(作者假设互作关系越保守,就越重要),并且想要搞清楚它们的互作关系。所以首先要构建出生队列,通过与成人菌群对比进行了alpha/beta多样性分析。观察到第36个月与成人的配置接近。随后,构建了一个年龄判别器,特征重要性分析找出了一批相关的菌,这些菌可能是在发育过程中起作用的菌。但是无法揭示这些菌之间的互作关系。接着,作者假设那些保守的共变化菌会起到重要作用。要想发现这样的菌,首先要寻找到系统趋于稳定的时间点。借助iPCA的方法,发现第20个月起,重要菌的结构已经不再发生变化。所以,接下来的目的就是通过第20个月起到第60个月的数据,找出那些保守的协变体(再一次强调,其基本假设是这些跨时间尺度的保守协变体起着重要作用)。因此,作者利用利用了20到60个月的taxa-taxa矩阵得到了一个叠加矩阵,这个叠加矩阵能反映系统稳定演化的过程中taxa的相关性(或者共变性)。通过对叠加矩阵进行2值化处理,可以只关注共变性处于top20%的菌。进一步对2值化后的叠加矩阵进行PCA,根据PC1上的载荷,提取了top20%系数的菌,从而得到了15个taxa,并构建了网络(网络节点稀疏且保守)。
Part II:其他队列的情况
接下来就是衡量找出来的ecogroup效果怎么样?因此,作者进一步提了3个问题:
1.ecogroup能否对孟加拉国的出生队列菌群结构提供一个概要性描述。
2.如果满足1,不同地理位置/人口特征的健康出生队列的ecogroup的改变是否遵循同一个pattern?(也就是说,它们演化的趋势是否一致)
3.第三点也是本文的初心:
(i)如果假设ecogroup可以作为有价值的正常人体的菌群发育marker(显然可以,ecogroup是随机森林重要特征的子集)
(ii)是否可以进一步利用这些taxa来表征菌群的受损程度,以及接受了干预治疗后的修复程度?
1.回答问题1
为了回答问题1,创建了3个矩阵,分别进行了PCA。
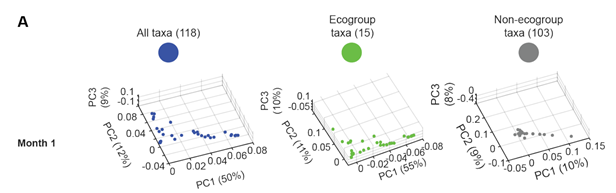
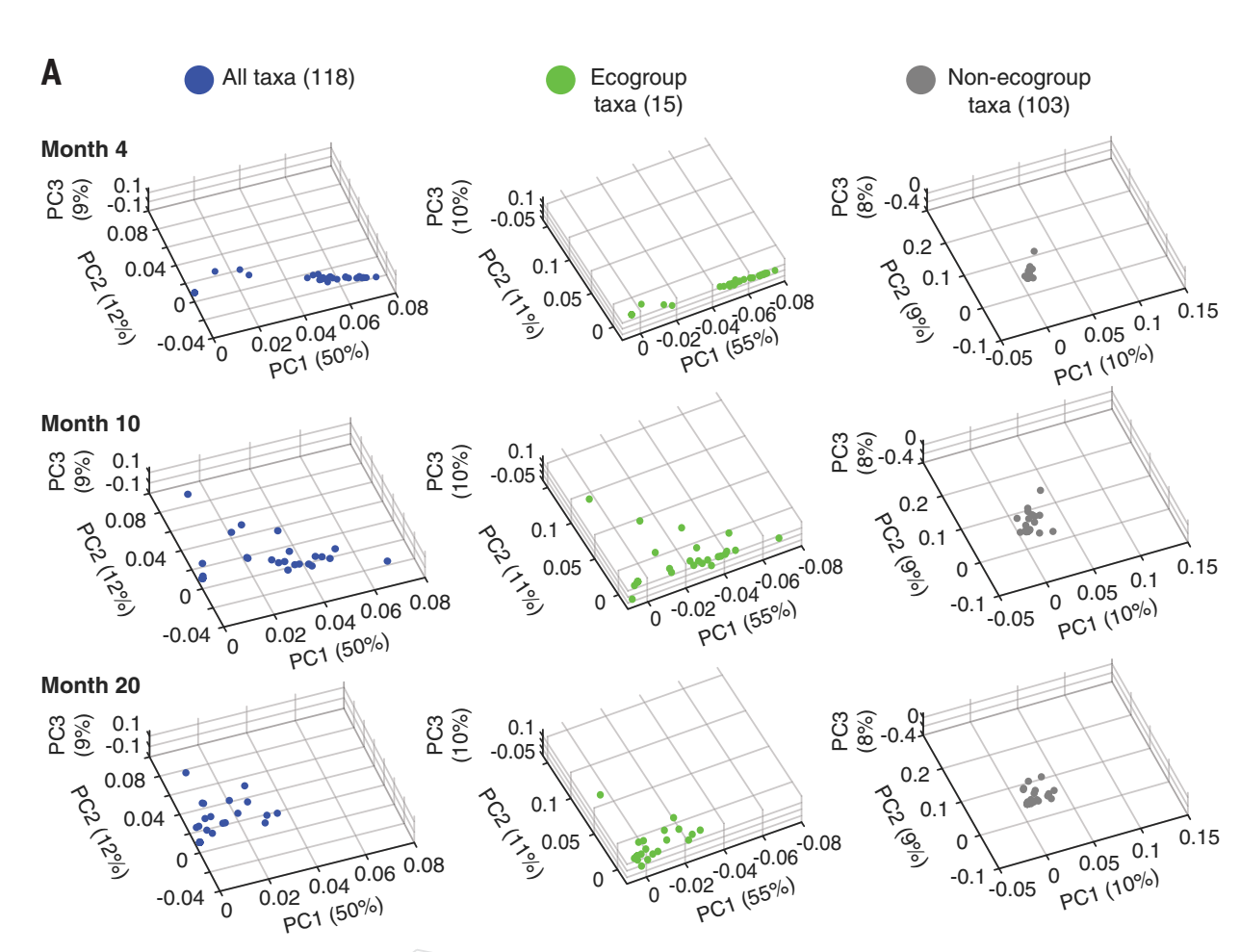
上图的描述:
1.第一个月,个体间的差异是比较大的(PC1可以看出);第4个月起,开始收敛。随着月份的增加,PC1从右向左收缩,PC2,3的差异也渐渐变小。
2.从第20个月份起,随着月份的增加,肠菌的pattern趋于稳定。(要注意,断奶期最长的一直到了23个月)
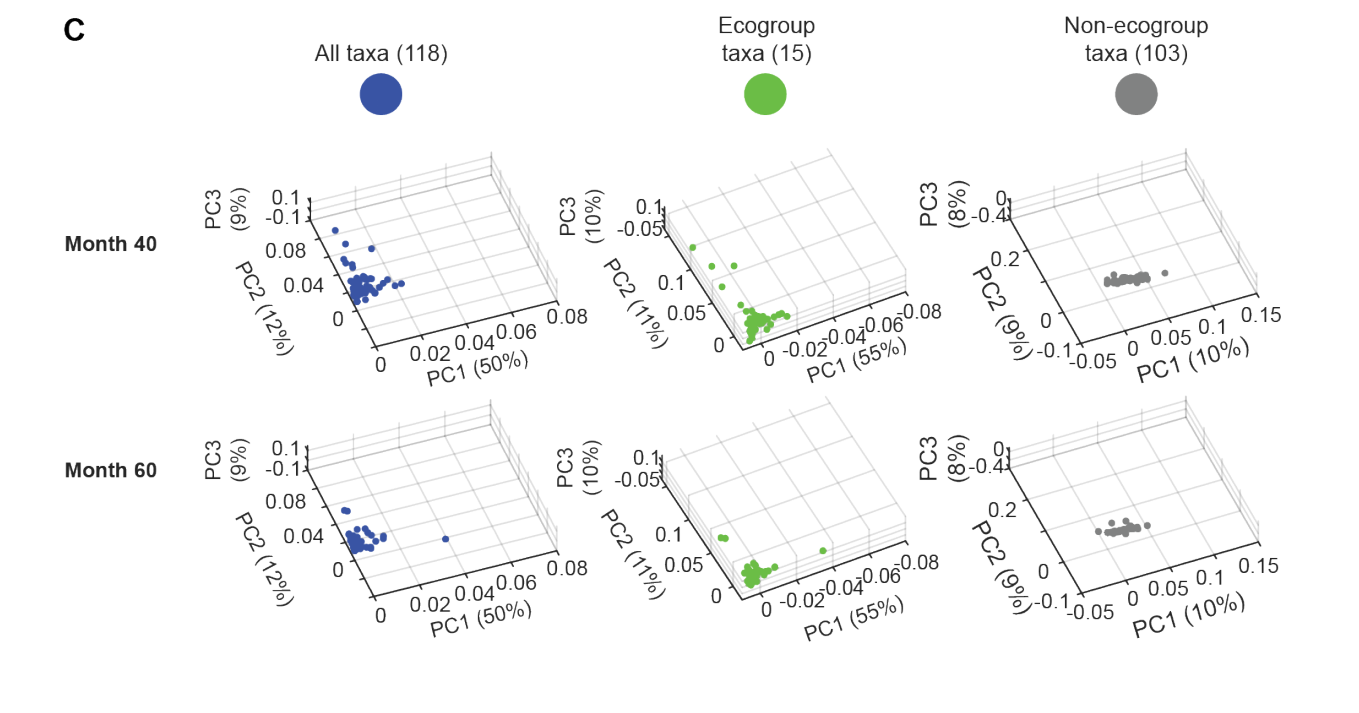
结论:eco-group确实能够起到一个概要性的描述。而non-ecogroup则很难起到一个好的描述作用。
2.ecogroup不同时间节点丰度可视化
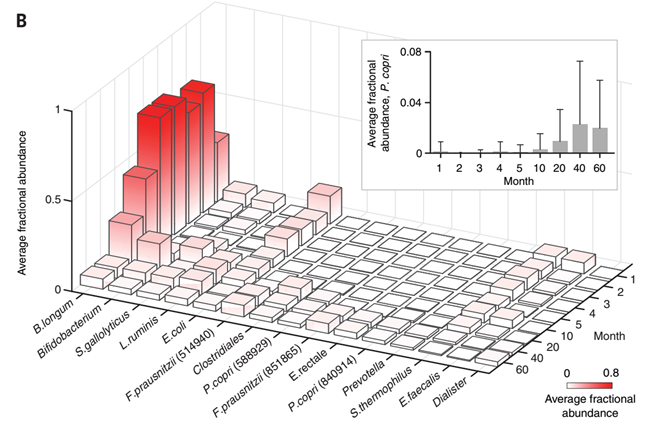
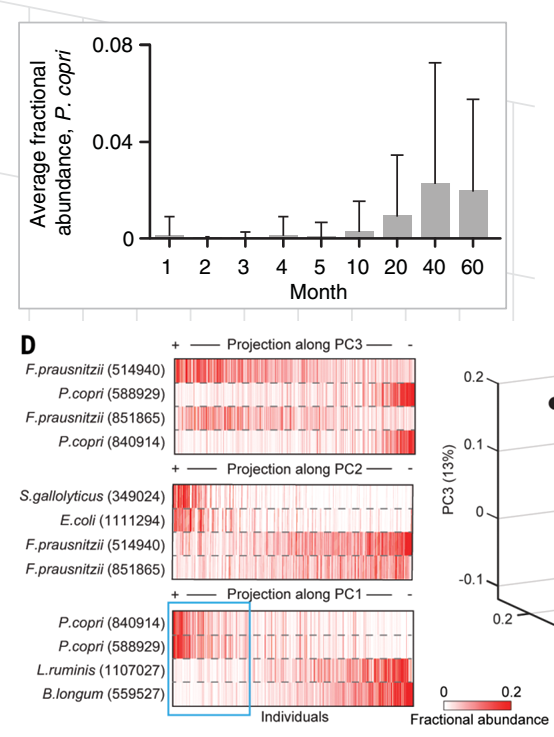
B图反映了15个ecogroup在不同时间点的丰度变化情况,特别强调了普式菌(P.copri),可以看出丰度很低。
作者特别强调了一些低丰度但变化大的重要菌如:普氏菌,在单独分析(指不考虑交互作用)时常常被忽略。但是作者通过保守性分析就可以揭露P. copri 这种低丰度但变化大的菌和其他菌的交互关系。
第二幅图(50 to 60月) 则特别强调了普式菌与长双歧杆菌(B.longum)的共同变化关系,可以看到是很强的负相关。
2.回答问题2
在其他队列(MAL-ED network 队列)中是否找到的ecogroup也会随年龄增长呈现同样的变化模式。
MAL-ED network 队列:中低收入国家。(出生前2年的队列,分别位于印度和秘鲁)
作者发现保守性taxa的前提是依赖于高时间分辨率的队列,而MAL-ED network 队列只到前2年,无法利用开发的workflow发现保守性taxa.
于是拿本文发现的15个ecogroup和在印度和秘鲁队列注释到的taxa去做PCA,发现了在随着月份的增加,呈现了同样的变化趋势(趋于稳定)。
结论:eco-group可以作为一个概要性描述。
3.回答问题3的第一部分
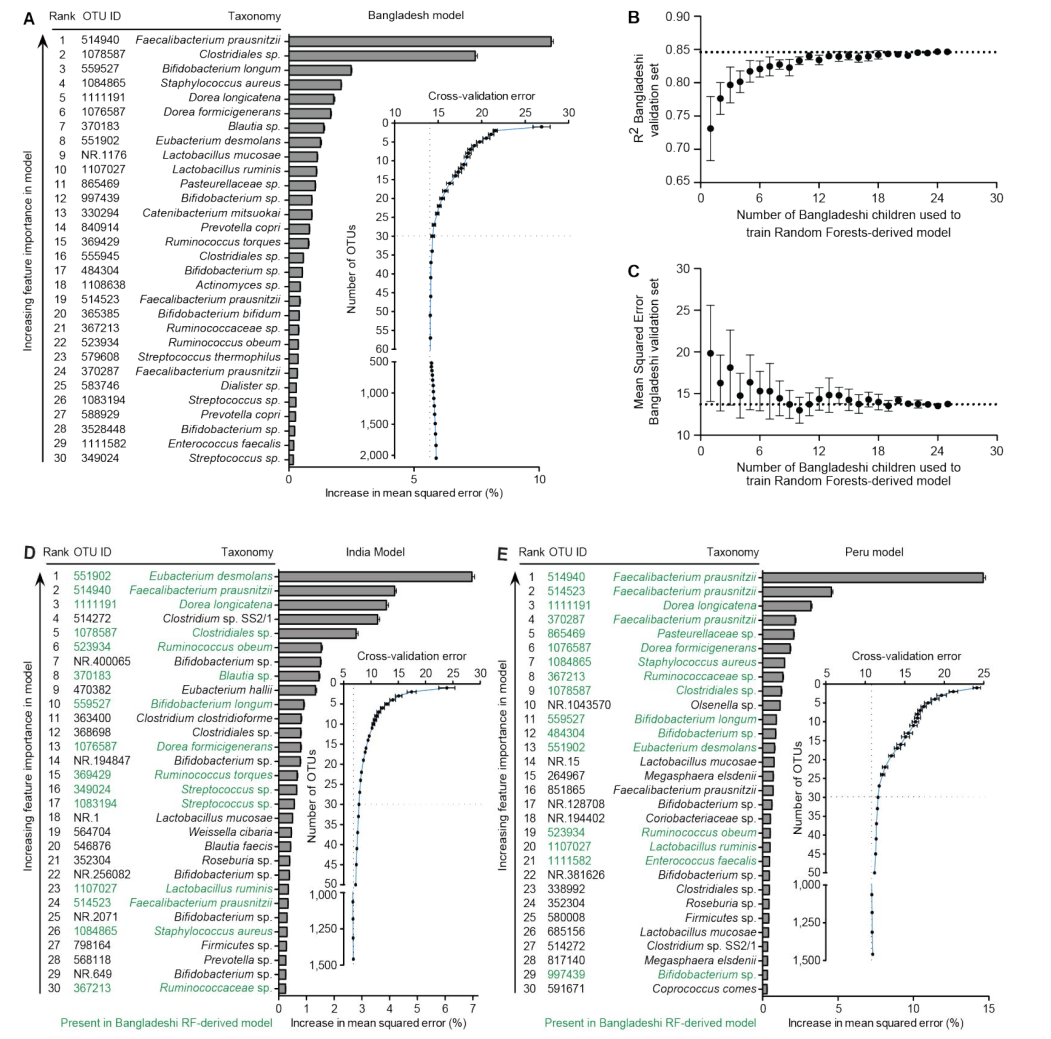
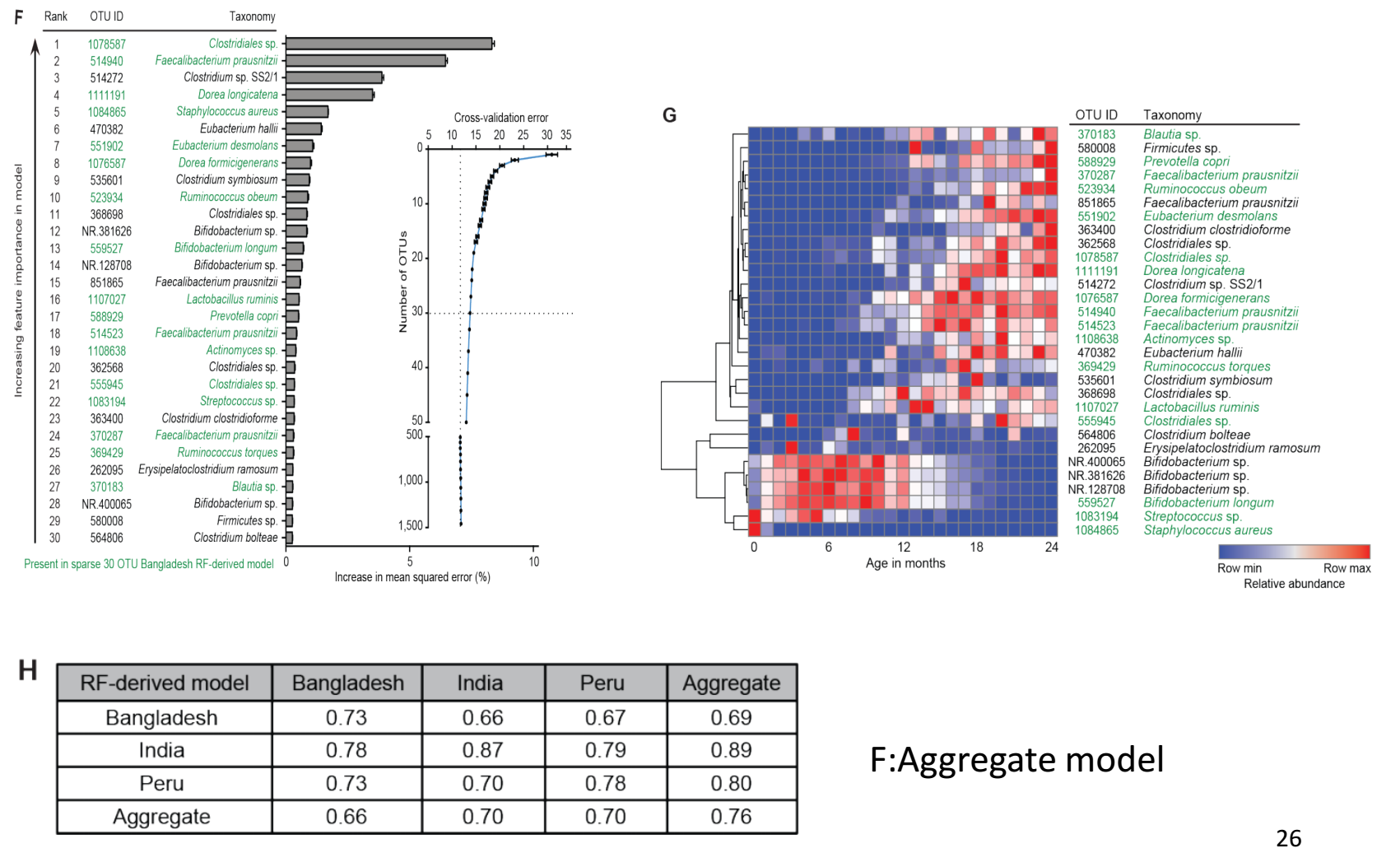
作者假设ecogroup可以作为有价值的正常人体菌群的发育marker。这个事情已经在孟加拉国队列中得到了验证。(构建了RF模型和ecogroup,RF得到了一批年龄判别相关的特征taxa,ecogroup则进一步探究了其中最为保守的15个marker的相互作用情况。)那么是否在印度和秘鲁两个出生队列中,ecogroup也存在同样的marker作用呢?
于是,作者在印度和秘鲁两个出生队列运用RF模型得到了验证。同时,还构建了一个整合模型(把3个数据集拿过来用)
Part III:急性营养不良儿童的ECO-GROUP
第三个问题的第二部分尚未得到解答:如果假设ecogroup可以作为有价值的正常人体的菌群发育marker,是否可以进一步利用这些taxa来表征菌群的受损程度?或者表征接受了干预治疗后的修复程度?所以,作者收集了MAM儿童的粪便样本,进行了随机双盲的临床试验,临床试验的结果发在另一篇science上。尝试回答该问题。
- 第二篇文章概要
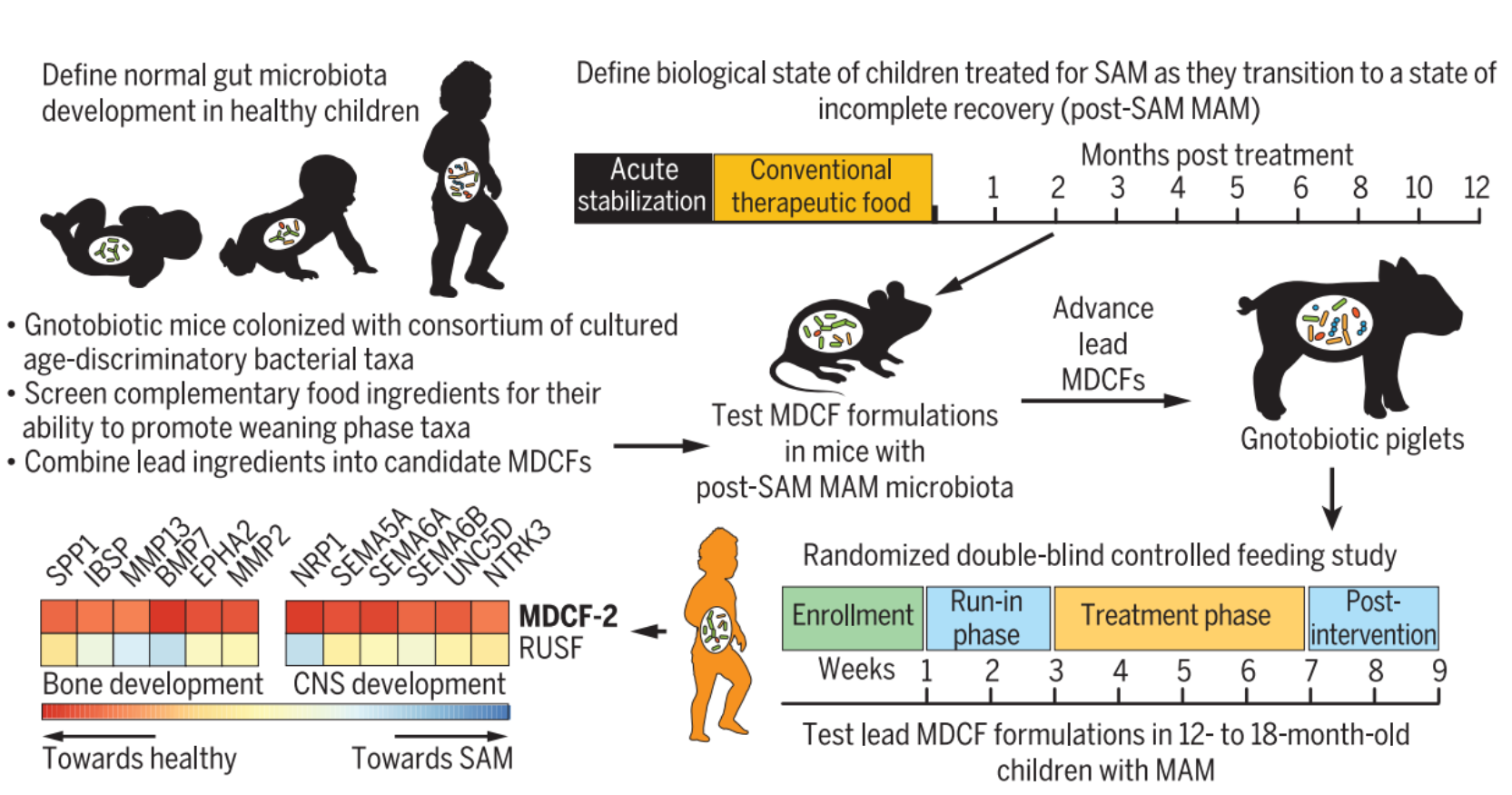
由于本篇文章的工作描述的是另一篇文章在执行过程中遇到困难后开发出来的方法,所以其实省略了第二篇文章怎么筛选得到治疗性补充食品的过程。一方面,他们首先是对三种易获取的食品在SAM上进行了临床试验,结果发现不是很好,没有完全修复,而是处于中间状态(Post-SAM MAM)。另一方面,通过前期SAM MAM样本收集,通过年龄判别模型,发现了MAM/SAM儿童肠菌年龄偏低。于是,从健康和MAM儿童中分离得到了一部分年龄判别菌群。随后,把年龄判别菌移植到无菌小鼠上,从候选食品中根据其对这些无菌小鼠的作用上进行筛选,标准就是能够提高MAM儿童肠道中没有的,但又能提高年龄预测值的年龄判别菌的丰度,这些食品作为进一步筛选的依据。进一步用一定的营养配比原则去设计食品组合。 最后在克隆了中间状态儿童菌群(Post SAM/MAM)的无菌小鼠,无菌小猪上进行了食品组合作用效果的动物实验验证。最终,筛选并得到了3个组合(MDCF1,2,3)加上一个RUSF对照 ,在MAM儿童进行了双盲随机的临床试验。
1.MDCF-1,2,3情况
3种最终进行随机双盲试验的补充食品
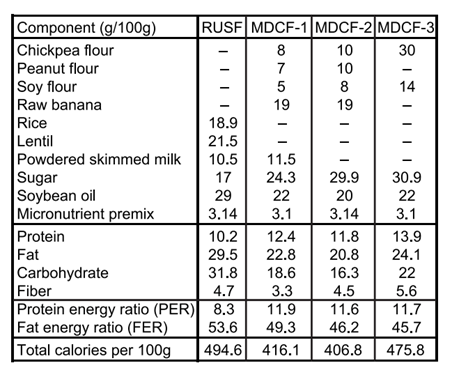
3种MDCF蛋白质能量比,脂肪能量比相似。食品的选取原则是根据当地的饮食习惯来筛选的。
MDCF1:
MDCF2:鹰嘴豆粉,花生粉,大豆粉,和香蕉,浓度比1,3高。
MDCF3:只有鹰嘴豆粉和大豆粉
RUSF:作为对照,缺乏所有四种成分,但能量密度,蛋白质能量比,脂肪能量比以及宏观和微量营养素含量方面与MDCF相似。
2.创建矩阵
为了衡量MDCF修复MAM儿童肠道的能力,创建了一个矩阵,矩阵包括:
SAM trial(接受了3种标准常规食品治疗)
MAM trial(接受了MDCF-1,2,3的治疗,时间选择:pretreatment and 2 weeks after treatment)
Age-matched healthy children
3组数据。并进行了可视化。
治疗情况可视化:
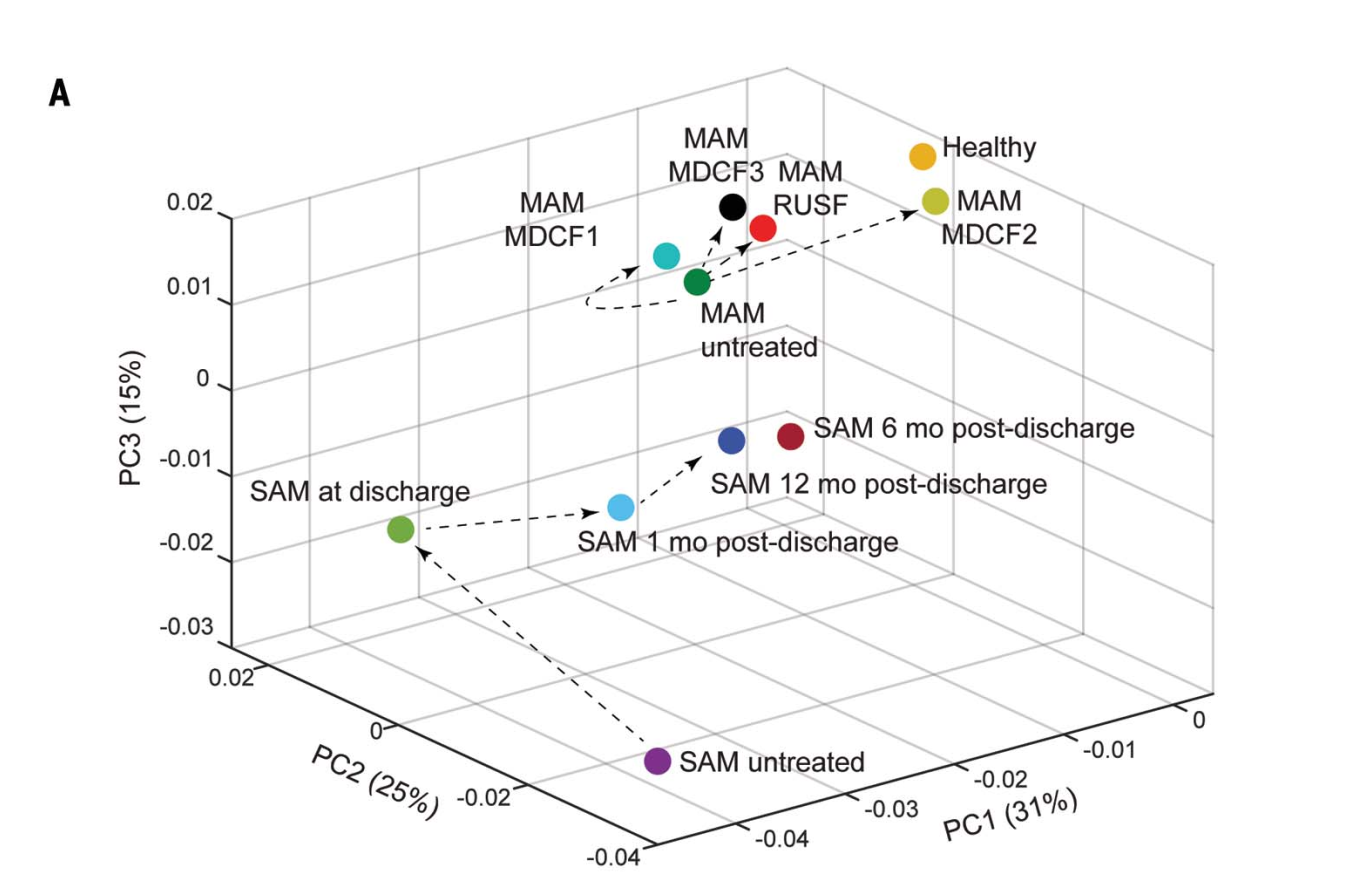
这幅图是整篇文章的精髓。
总体上,可以看到接收治疗后都离健康原来越近了。
1.SAM接收标准治疗的处于不完全修复状态,出院后1 6 12月都有改善,但是离健康还有一点差距。
2.MAM:MDCF-1/3,RUSF的治疗恢复情况相似,而MDCF2则非常接近healthy.
3.载荷分析
进一步进行了载荷分析,发现了一些系数较大的菌:B. longum, S. gallolyticus, and E. coli。
分析了载荷情况,根据投影图结果和载荷系数,作者认为还应该进一步减少B. longum S. gallolyticus的丰度,进一步增加P. copri 的丰度,才能到达正常人的位置
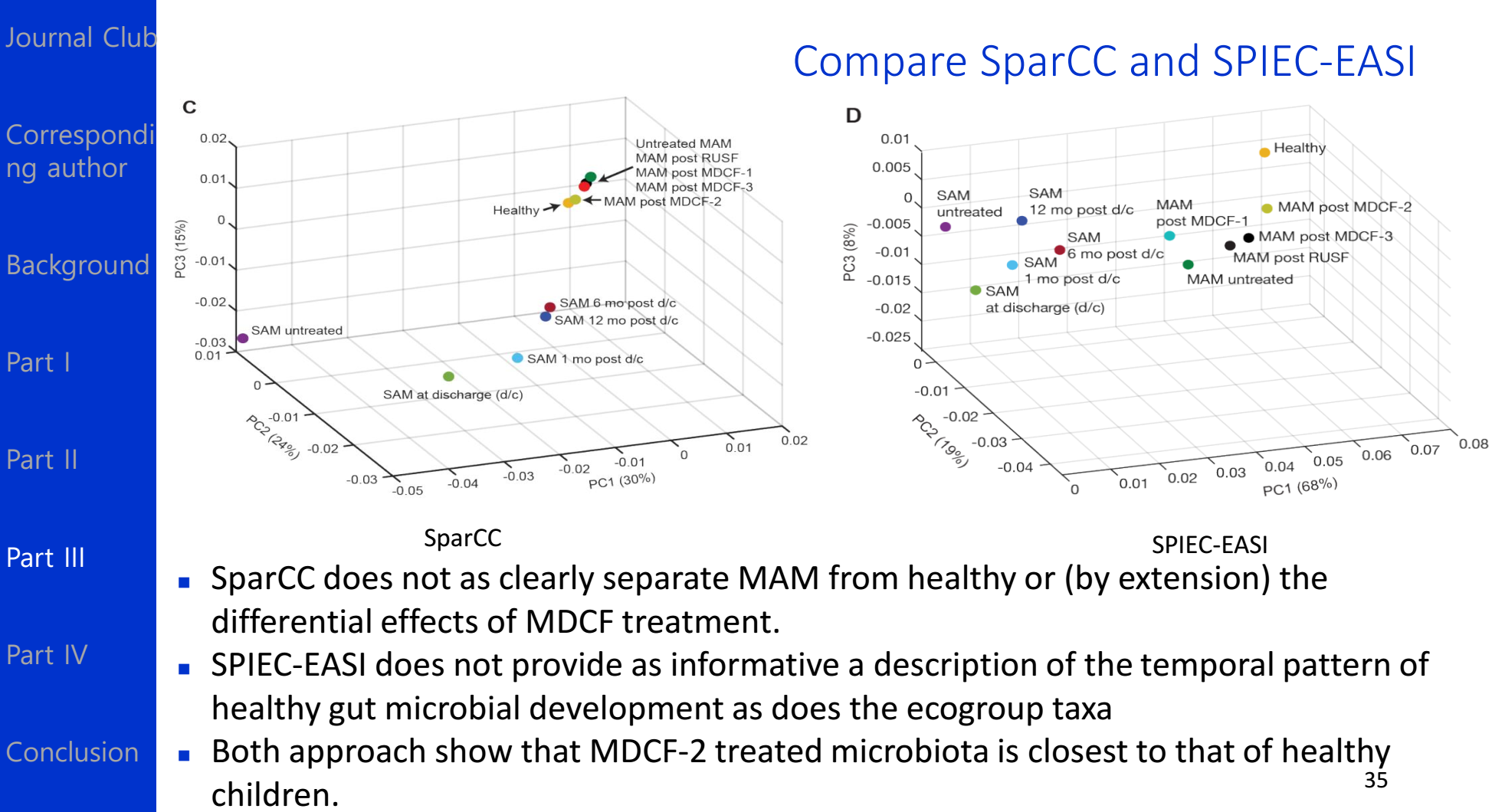
4.与SparCC和SPIEC-EASI 的比较
为了说明自己的方法好,与2个流行的方法进行了比较。2者为横断面研究设计,改造了一下。
比较3者发现1.保守元素的能力2.衡量修复程度的能力。
结论:
1.SparCC没有明显区分MAM和健康人,MDCF之间也没很好区分。
2.SPIEC-EASI没有提供有价值的描述,PC轴的排序比较乱。
3.但是两种方法都表明 MDCF2与健康人最接近。
4.对生理发育指标的改善情况(另一篇paper中提到的)
在Paper2中(见下图,GO富集分析结果),还提到MDCF2的血浆蛋白组测序表明,MDCF2改善了健康装药,包括一些新陈代谢的生物标志物和调节剂、骨骼生长、中枢神经系统发育和免疫功能的改变。
Part IV:在无菌小猪上观测肠道菌群随饮食的变化
进一步提出一个问题,这一步感觉可以写第二篇文章了。观察到的这些结果使作者好奇,这种菌与菌交互的特性是如何随着断奶期的时候饮食的改变而发育的。在人身上由于不同家庭的断奶期不同,没法控制变量进行做实验观察,因此,作者在germ-free小猪上进行了试验,目的就是为了尽可能还原健康人在断奶期期间随着饮食的变化,ecogroup交互以及丰度变化的过程。因此,移植了ecogroup到猪的肠道,并进行了不同时间节点的取样观察。选择猪的目的是因为生理和代谢特性与人类相似。

1.实验设计情况,见下图:
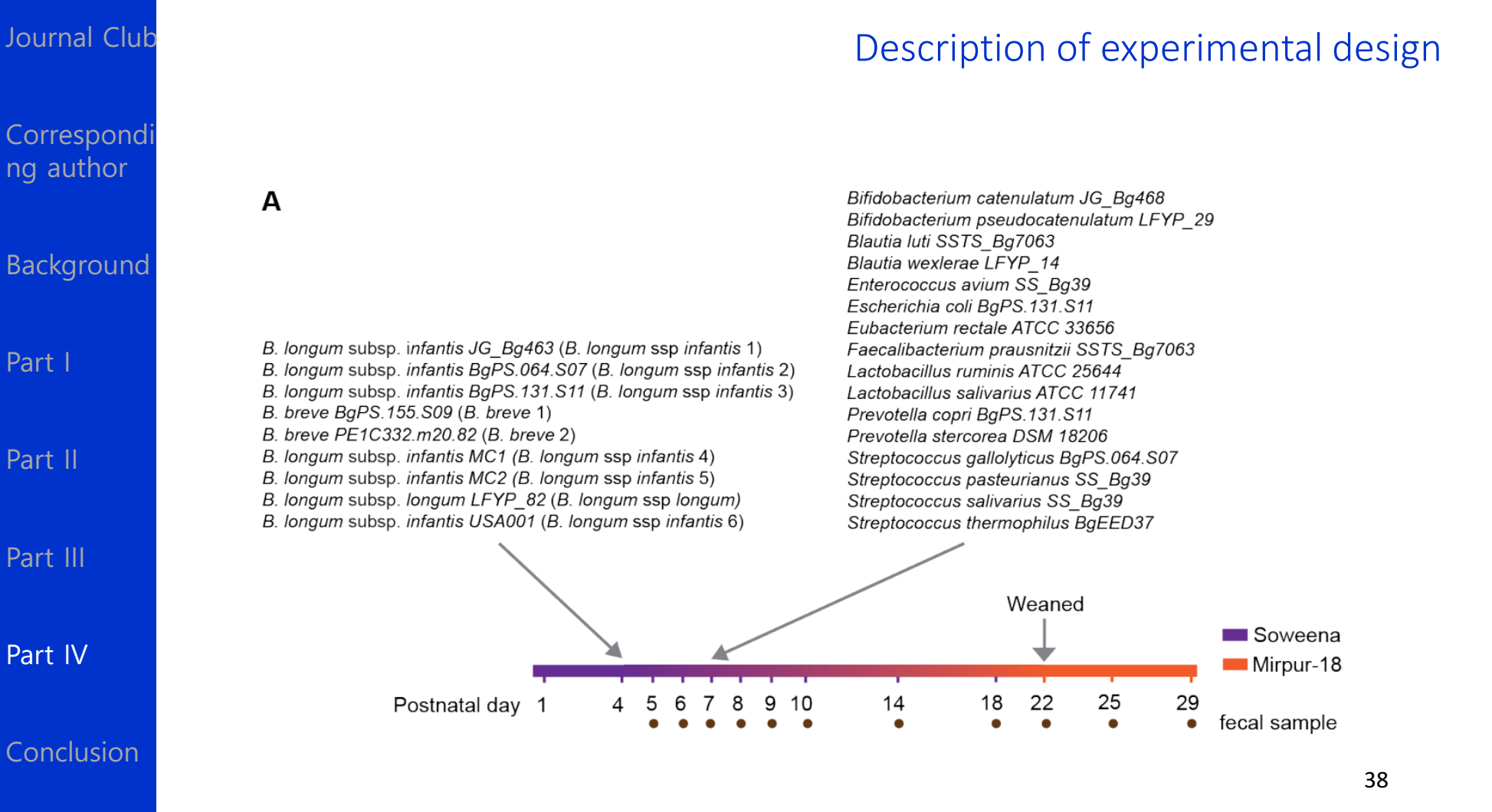
before day4:小猪只喝受辐射过的牛奶。
day4:此后,通过口服了7株可培养的B.longum (全是长双歧杆菌的菌株,B. longum subspecies infantis and one as B. longum subspecies longum )还包含了2个Bifidobacterium breve strains作为比较者,用于描绘有助于健康的要素。
day4 起,Mirpur-18 逐渐添加mirpur-18(某18月小孩食用的套餐)
day5-22起,mirpur-18浓度递增;牛奶递减。
day7:进一步移植了16株菌,一共13个物种,全部在ecogroup中。
day22:完全断奶,仅喂食mirpur-18
day29:安乐死。
2.宏基因组测序表征菌群丰度变化
为了定义在断奶期,ecogroup菌株与健康相关的特征(感觉就是一些代谢与生合成的通路相关基因),5-29天按时间节点直肠擦拭进行了宏基因组测序,在最后一天沿着肠道的长度进行擦拭测序。结果:
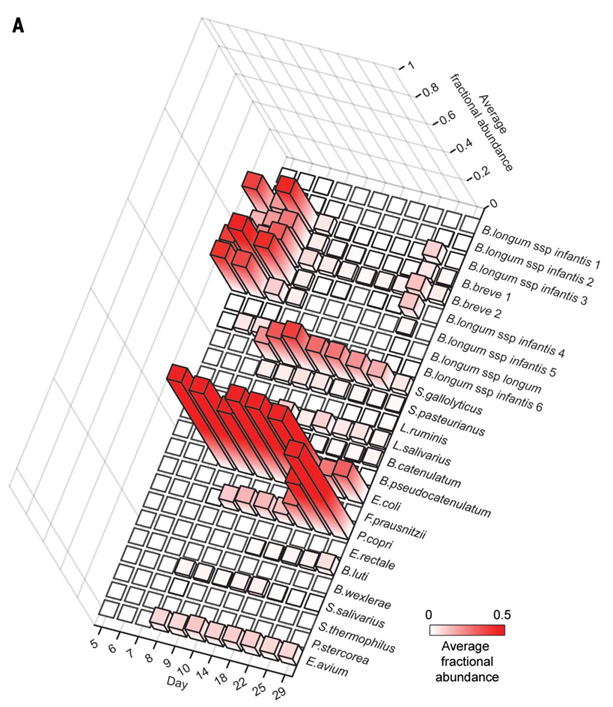
图A:
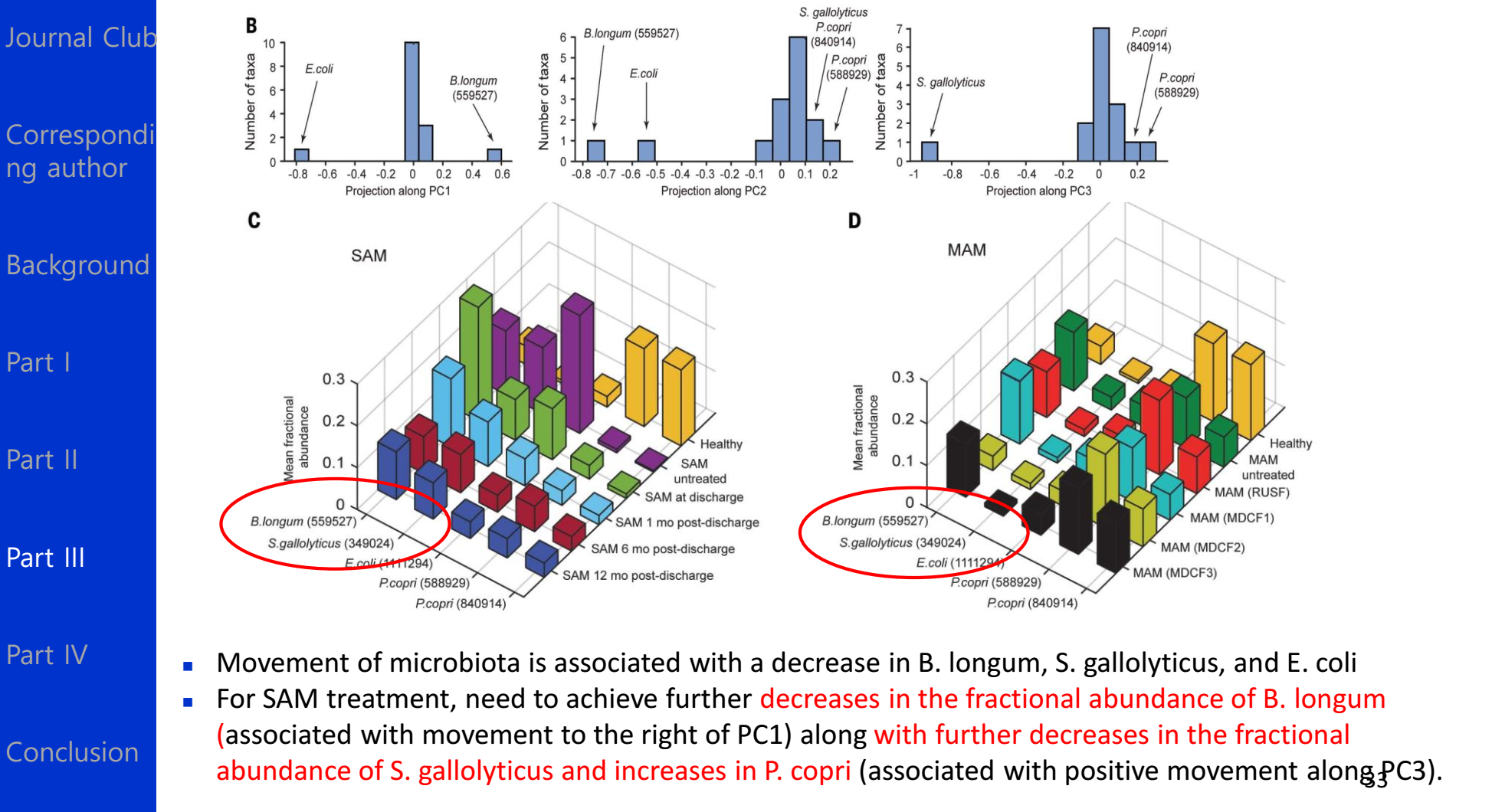
1.B. longum 在第7天(定制了16株保守菌后)后显著减少
2.8-22天断奶期,S. gallolyticus, E. coli, E. avium,L. salivarius, and P. copri呈现了不同的变化模式。
3.完全断奶期(day22)P. copri 显著增加,最后成为盲肠,结肠和粪便的主要成员(day29安乐死后看的)。
值得注意的是,B.longum与P. copri的关系在猪中与在人中的变化关系类似。(都是强负相关)
3.宏基因组测序表征基因组特征(与菌株健康程度相关的特征)
根据mcSEED代谢注释数据库注释结果对菌株的以下能力进行了预测:
1.合成氨基酸,B维生素的能力。
2.利用碳水化合物的能力
3.产生短链脂肪酸的能力
预测结果0,1表示营养缺陷型或者原养型。
热图可视化:对结果进行了热图展示。8-18天反应的是断奶期,22天完全断奶。
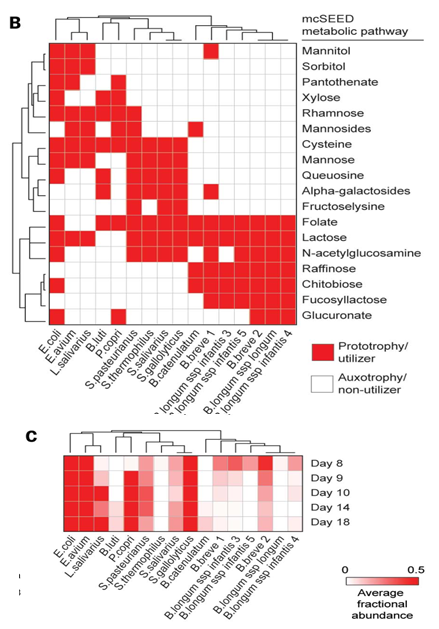
B:代谢通路热图
C:菌的丰度在断奶期的变化情况。
4.宏转录组测序

作者定义了一个富集矩阵,上图表明了产生流程。过程类似于KEGG富集分析。中图是富集矩阵可视化,右图是P. copri 作为了参考系(可以看到这一列是0),因为它在第29天有着最高的丰度。热图可视化中,富集计数比它大的是红色,小的是蓝色。
对富集矩阵进行PCA:
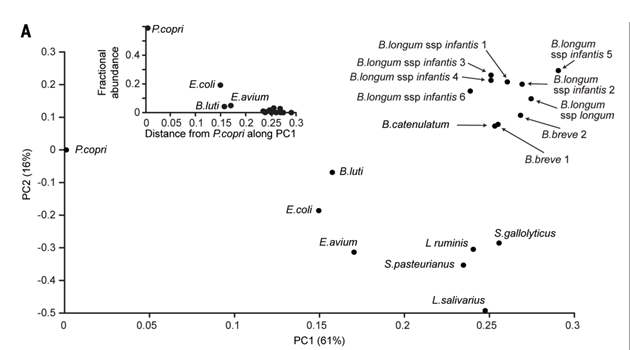
发现Bifidobacterium的代谢模块相似,因此 Bifidobacterium聚集到了一起。
还发现投影的PC1(代表了代谢水平)与丰度有相关。
为了进一步探究哪些代谢模块对不同的丰度起到贡献,作者需要使用一个能把富集矩阵的代谢模块(行)与列(菌株)连接起来的方法。他们用了SVD。
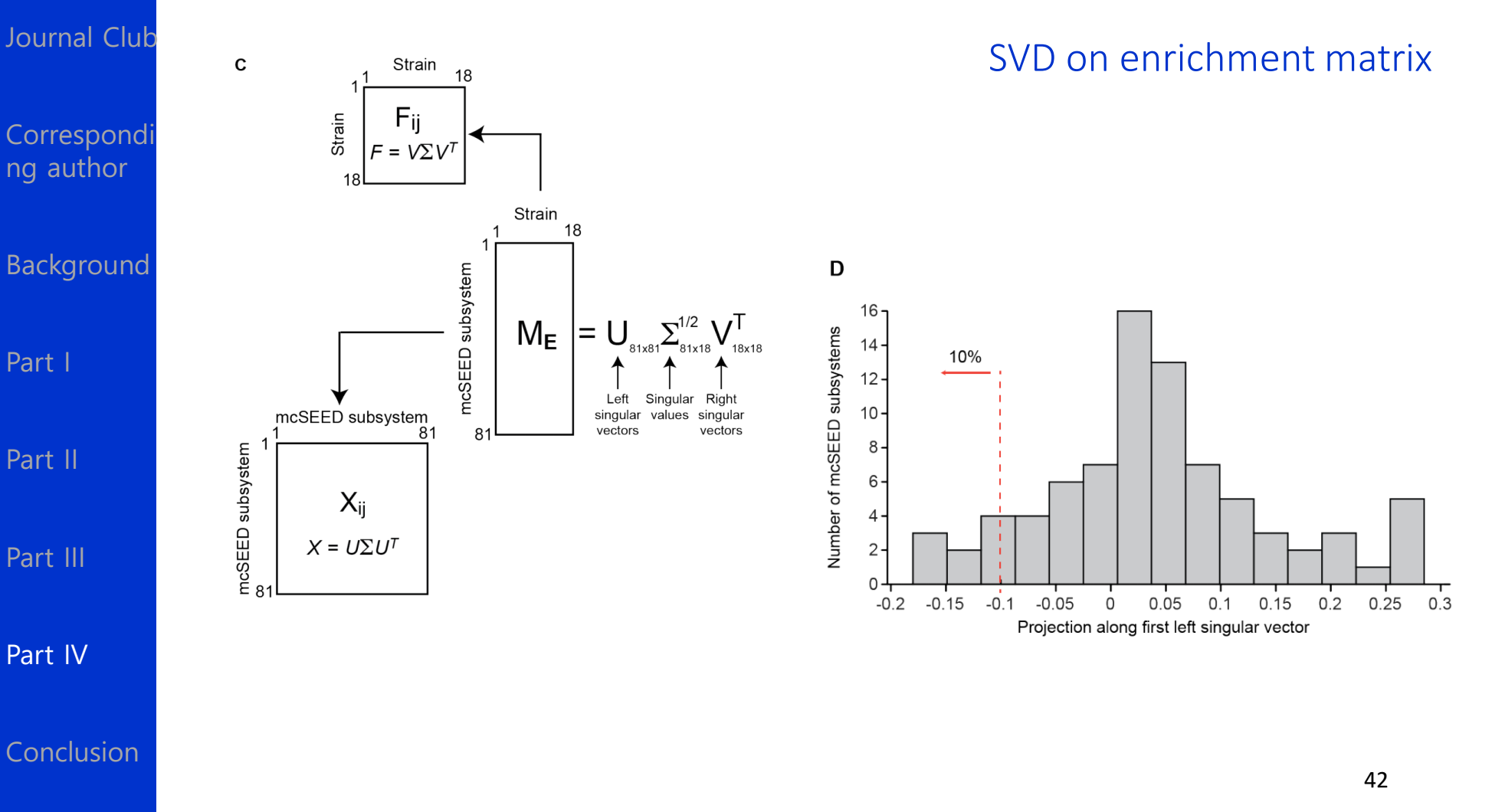
(因为SVD能够分解出一个左奇异向量矩阵和一个右奇异向量矩阵,恰好可以用于表示代谢模块(行)与列(菌株),而左奇异向量矩阵就是PCA中对应了特征向量矩阵。
关于SVD:也是一种降维的手段,与PCA由异曲同工之妙,只不过SVD能够同时对行和列进行表示,而PCA不行。表示的意义是人为赋予的,但是往往都很有效。
表示的观点出发:每一个菌可以由若干代谢模块表示。每一个模块由可以由不同的菌进行表示,所谓表示,就是通过不同的系数进行线性组合。具体一点:左边这个U矩阵的每一列可以表示一个文档(对应一个模块,都是81*1的向量),而右边V矩阵的每一行对应一个单词(对应了该模块可以由不同的菌表示,一共18个菌,因此是18 x 1的向量)
如果对中间的奇异值矩阵按照大小排序,就可以求解出方差最大的投影
作者沿着第一左奇异向量的方向(表示方差最大的模块)进行投影,即这个模块是最能够区分开不同菌株的模块。
再看下图:
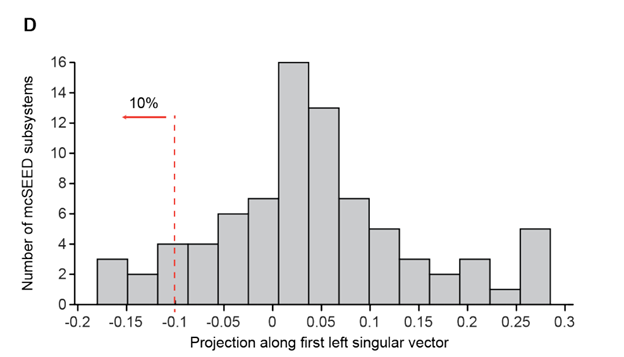
他们做了SVD以后,发现在该第一方差模块上投影贡献最低的都是一些双歧杆菌,在红线左侧的表示这个投影的系数相对于P.copri(参考系)来讲,非常低。于是,进一步拿了前几个方差模块(左奇异向量进行投影),发现下图:
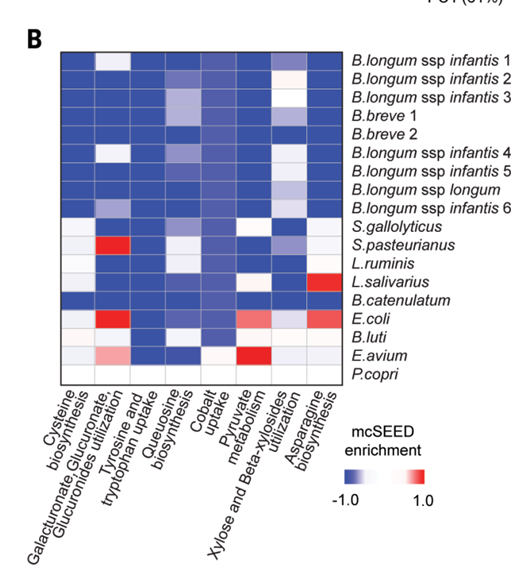
他们发现:相对于普式菌来讲,最能够区分开它和双歧杆菌的特征是一些明显的降低或缺少了表达量的通路:
1.生物合成:半胱氨酸,酪氨酸,色氨酸和天冬酰胺。
2.利用碳水化合物:木糖和β-木糖苷加半乳糖醛酸/葡萄糖醛酸/葡糖苷酸
3.辫苷的生物合成
4.钴胺素生物合成相关的钴的摄取
而且上面者4个通路可以完全区分开双歧杆菌和其他细菌。见上图。
这种完全区分开的特征通路促使作者进一步在基因组水平上看这些系统是否完整。下图展示了双歧杆菌的代谢通路情况。
完整的:酪氨酸,天冬酰胺和色氨酸生物合成
没有的:半乳糖,木糖和葡萄糖醛酸苷的途径;和B-维生素合成的queuosine途径和钴胺素
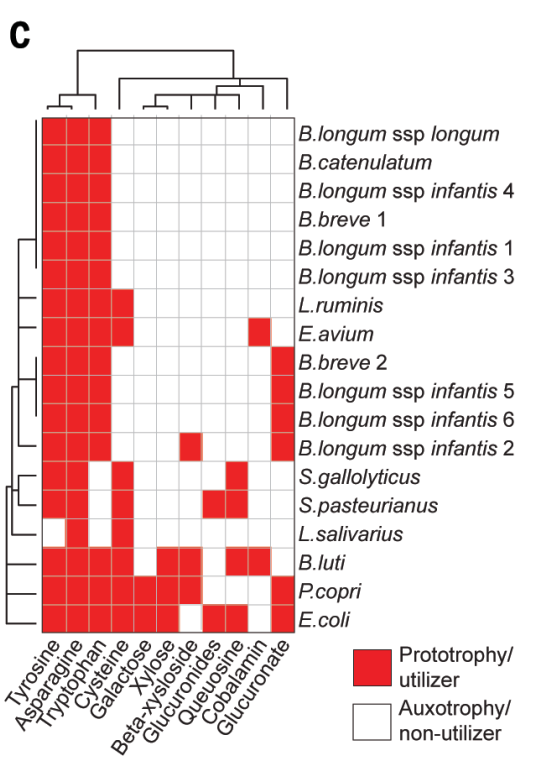
这些结果表明,所检查的双歧杆菌菌株的基因组特征限制了它们繁盛能力,这是由在Mirpur-18的饮食和其他菌构成的环境共同作用造成的结果。相比之下,P. copri和其他生态群体菌株含有并表达这些代谢途径为其维持健康的重要性提供了支持在这些条件下。
因此,作者得出结论:这种降维方法提供了一个合理的视角解释:通过饮食的营养干预,某些营养靶向ecogroup的这些通路,有可能会对儿童的微生物发育造成扰动。
结论
1.发展了一种统计方法,能够识别发育中的微生物群的保守性成分。
2.该方法能够区分开SAM/MAM/Healthy儿童,以及衡量损失修复程度。
3.在无菌猪上的实验观察,证明了饮食,某些菌的在某个时间点上突然出现的高丰度改变(P.copri)会共同影响其他菌的出现与否(B.longum)。
以上3点共同表明,本文发展的方法能够适用于其他纵向数据。
4.通过在高维数据中嵌入一个稀疏的保守性协变网络能够表征一个优雅,鲁棒而又允许适应的自然生态系统。
上一篇: Introduction to MultiDimensional Scaling
下一篇: 深度学习应用系列——NeuSomatic:鉴定体细胞突变