DADA2
题目:dada2: high-resolution sample inference from illumina amplicon datas
作者:Benjamin J Callahan1, Paul J McMurdie,Michael J Rosen, Andrew W Han, Amy Jo A Johnson2 & Susan P Holmes1
杂志:Nature methods, published: 23 MAy,2016
DADA2是一个用于建模和纠正Illumina扩增子测序错误的算法(https://github.com/benjjneb/dada2)。
DADA2准确地推断出样品序列,并解决了仅1个核苷酸的差异问题。
在一些模拟社区中,与其他方法相比,DADA2识别出更多的真实变异并输出了更少的伪序列。
- 介绍
扩增子测序中一个很重要的问题是如何从在带有测序错误的情况下(很显然,基于OTU的方法没有考虑测序错误的因素),从而解析真正的生物学变异。该挑战促使人们开发了扩增子的错误校正方法。
在DADA2发表以前,尚无针对illumina平台的错误校正方法。
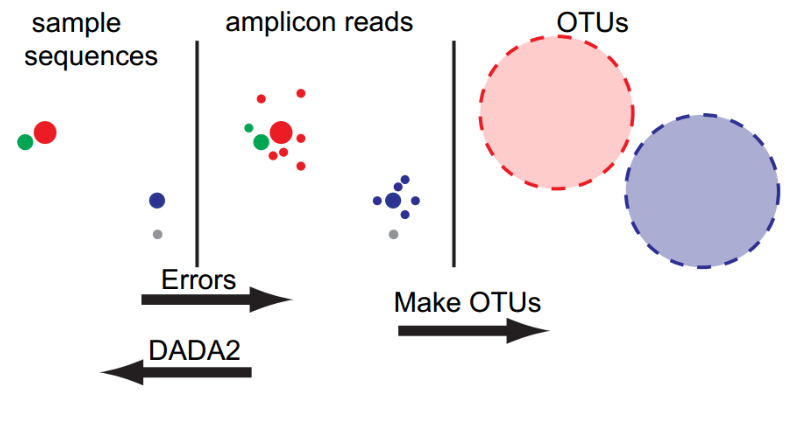
- otu方法的两大致命问题
目前,基于illumina平台扩增子测序错误的解决办主要是通过:先质量过滤,随后构建OTU来解决。
然而:
1.简单的根据相似度进行聚类会错把测序错误当做生物学变异,因此OTU的方法并没有进行假阳性推断。
2.此外,OTU方法没有利用测序错误模型,从而无法进行精细尺度变异(Fine-scale variation)的解析。
- 精细尺度变异与DADA2
精细尺度变异(Fine-scale variation)的好处很多。最浅显的:可以将某些菌株中的致病菌与普通菌株区分开,并且可以在复杂的微生物组相关疾病中提供更多的临床相关信息。
DADA2完整的扩增子工作流程:过滤,重复数据删除,样本推论,嵌合体识别以及成对末端读段的合并。
- 算法流程
1.根据error model计算error rate,error rate是量化了测序序列i产生自样本序列j的概率,error rate是序列碱基组成以及碱基质量的函数。
2.理论上,read i产生自read j的数量可以描述为泊松分布,期望值lambda= error rate x (# of read j)。如果i真的来自j,那么观测值(即,i的丰度)应当与期望值相等。根据这一事实,可以进行假设检验。计算的p-value记作abundance p-value.
3.abundance p-value被用于作为迭代partition算法的是否要继续迭代的阈值标准;当所有abundance p-value均满足阈值要求时,我们可以认为:同一个partition下的序列都产生于该partition的central sequence。最终,推算出来的sample组成就是每一个Partition中的center 序列以及对应的丰度。
【注:一个partition可以看做一个cluster,降噪体现在最后是以center序列作为partition的代表,这样就消除了partition内的其他序列由于测序原因而引入的错误。】
算法细节
error model
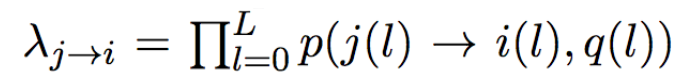
dada2假设在reads内的测序错误以及reads之间是相互独立的。
在这种假设下,样本真实的序列j产生测序序列i的错误率(error rate,即公式中的Lambda)可以描述为:每一个碱基替换概率的累乘函数。此函数与替换的碱基类型(也包含不发生替换的情况),碱基测序质量得分有关。
例如,p(A→C, 35) 表示该碱基测序得分是35分的情况下,从A替换为C的概率(显然是一个很小的值)。
当然,在实际算法运行过程中,显然在起始条件下并不知道哪一条序列是真实的样本序列j,因此,采用启发式的策略,人为的把在一个partition下丰度最高的unique read假定为真实序列j,其他unique read与j进行比对,并计算相应的lambda(j->i),(具体参考partition一节)。
如果序列j和序列i很相似,error rate应该是一个很接近1的值。如果两者匹配不上,那就直接把error rate分配为0。
abundance p-value
如果测序的错误在一段reads上是独立的,那么,真实的序列j产生测序序列i的数量应当服从泊松分布,其中测序序列i的期望值应当为:
期望值=error rate x 期望的样本真实序列j数量
error rate可以从error model计算得到
期望的样本
真实序列j数量显然无法得到,但可以采用启发式算法进行迭代求解。(参考下一节)
根据这个期望值以及实际得到的观测值,我们就可以利用假设检验判断read i是否来自于序列j。
因此,进一步进行假设检验:
H0:测序序列i的观测丰度 = 理论期望值
H1:两者不相等
p-value可以用于衡量一条测序序列i产生自真实的样本序列j的可能性大小。
如果P-value很大,则无法拒绝原假设,我们就认为测序序列i是来自序列j,即i和j最后被划分为同一个partition;
计算p-value:
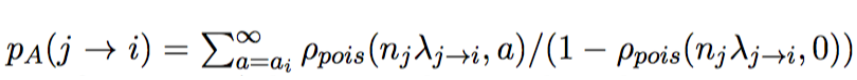 其中:
其中:
j是假设的来自样本的真实序列,i是测序序列,n_j是期望的样本真实序列j数量,lamda是error rate,a_i是read i的实际观测值。
n_jxlambda就是真实的序列j产生测序序列i的数量的期望值。
个人的思考:
p-value的定义:the probability we get this sample and a more extreme sample under H0.
因此,根据p-value的定义:在本例子下,我们的p-value可以计算为:序列i的丰度观测值a_i以及更多的情况下的概率。
所以,原论文中Then, conditional on i being read at least once, the abundance pvalue is the probability of seeing nj or more identical reads 的表述应该是错误的。
公式中的分母:
\[\frac{1}{1-\rho_{\text {pois }}\left(n_{j} \lambda_{j i}, 0\right)}\]可以这么解释:
除以1- p(0)是为了剔除掉观测值为0的情况(显然,观测值不可能为0)。由于概率分布之和为1,因此除以1-p(0)进行重新归一化。
显然,p-value越小,越表明现有的reads丰度无法被error model所解释,也就意味着这个测序序列i不是来自于该真实序列j(再一次强调,在实际算法运行过程中,显然在起始条件下并不知道哪一条序列是真实的样本序列j,因此,采用启发式的策略,人为的把丰度最高的unique read假定为真实序列j,其他unique read与j进行比对,并计算error rate 以及进一步根据error rate计算abundance p-value)
注意:
1.p-value显然需要一个阈值,用于判断是否显著,这个值通过OMEGA_A 参数进行设定,默认:1e-40。
2.启发式算法采用迭代策略,直到所有partition内的abundance p-value都大于阈值。
3.singleton 的p-value被设定为1,并且不会被error model判断与真实序列j的一致性。这意味着,singleton无法构建partition(具体参考partition:分配算法一节),因此DADA2不会推断singleton
partition:分配算法
前面一直提到:在实际算法运行过程中,显然在起始条件下并不知道哪一条序列是真实的样本序列j,因此,采用启发式的策略,人为的把丰度最高的unique read假定为真实序列j,其他unique read与j进行比对,并计算error rate 以及进一步根据error rate计算abundance p-value)。
具体的分配步骤:
1.扩增序列被进行去冗余,从而得到若干unique reads;每一个unique read可以根据原始reads数量换算成丰度,以及求取平均质量。(平均质量需要被用于error model)
2.将所有unique reads分配到一个partition中,将丰度最高的read分配为中心。
人为将丰度最高的reads作为中心,并假设为真正来自于样本序列,个人认为这是因为:
由于聚类过程是根据完全一致性进行unique reads划分,这个过程很显然,由于测序的错误,许多本是来自于同一个样本序列的reads会被划分为不同的unique reads;而丰度最高的unique reads显然可信度是最高的,因此也可以认为这个unique reads与样本真实序列是最近的。
其他所有unique reads与中心read进行比对,计算error rate: lambda(center->each unique read),以及进一步计算abundance p-value。
3.如果最小的p-value(经过bonferroni校正)小于用户设定的阈值OMEGA_A (根据经验设定,默认是),这就表明算法认为该partition中仍然存在着不是来自于真实序列(partition center)的测序reads;此时,将继续创建一个新的partition,并把p-value最小的reads分配为新的中心,其他所有unique reads与新的中心逐一进行比对,一旦发现新的中心更可能产生该unique reads,那么就把该unique reads划分为新的partition。
4.第3步不断进行迭代,直到所有的p-value都大于OMEGA_A 。此时,可以认为,每条序列已经被划分为最可能产生它的partition中。
最终,根据1-4步推算出来的sample组成就是每一个Partition中的center 序列以及对应的丰度。(降噪体现在最后是以center序列作为partition的代表,partition内的其他序列由于测序错误的原因,最后直接被忽略了)
error model的参数
前面提到的error model中的替换概率问题,error rate的计算依赖于每一个碱基替换的概率,而每一个碱基的替换情况由于实际碱基测序质量有关。
因此,实际上存在16x41个参数
41代表了所有质量得分情况(1~41),16是4种碱基替换情况,见下图:
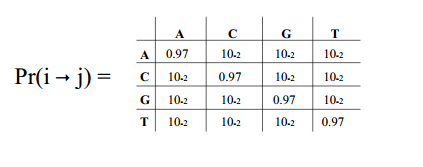
因此DADA2提供了3种选择:
1.基于用户的实际数据进行参数估计(利用dada2内置算法selfConsist )
2.(默认)单独对每一种替换,进行基于weighted loess 的拟合
regularized log (observed mismatch rates ) ~ weighted loess function(mismatch base quality)
3.用户自己定义一个参数估计函数。
测评
- benchmark
与4种算法进行了比较:
UPARSE
MED
mothur(平均链接)
QIIME(uclust)
- 模拟社区
在模拟社区进行了测评:Balanced,HMP和Extreme
平衡的社区包含在理论上相等的频率:57种细菌和古细菌。
HMP社区包含21个细菌在名义上相等的频率17,
而极端群落包含27个细菌菌株,其频率跨越五个数量级,在整个序列区域上相差至少1个核苷酸(nt)。
- 测评指标
将输出序列与组成这些模拟社区的参考序列进行了比较,根据算法结果,输出序列被分成了3种:
Exact hit: blast比对完全比对上了参考序列。
One-off:存在一个Mismatch 或者 gap。
Other:其他情况。
sensitivity :定义为Exact hit的比例。(需要注意的是:由于精细尺度变异广泛存在于模拟社区中,因此有些参考的菌株在16s序列中包含了多个可以区分的序列变异。)
DADA2软件流程
分裂扩增子去噪算法(Divisive Amplicon Denoising Algorithm.)
考虑了测序质量,无需参考数据库。
核心降噪算法是基于Illumina测序错误模型。
1.过滤: fastqFilter()
与usearch fastq_filter命令类似
此函数将序列修剪为指定的长度,删除短于该长度的序列,并根据歧义碱基的数量、最低得分、reads中的期望错误进行过滤。
fastqPairedFilter() 实现了同样地功能,只不过输出的是前、后端都通过过滤的reads。
2.重复删除 :derepFastq()
derepFastq()导入一个fastq文件并输出重复序列的唯一序列及其丰度。
derepFastq()还会输出一致序列的碱基质量得分(通过计算一致序列中对应位置的碱基质量平均值。)
该质量得分用于error model的dada函数。
3.降噪:dada()
细节在前面已经描述。
4.去除嵌合体:isBimeraDenovo()
去除嵌合体最好dada()之后,推断样本系列之前进行。
而且,最好不要使用独立的嵌合体去除算法(因为当前大多数独立的嵌合体识别程序,在识别与其他更丰富序列时更加保守,因为此类序列有望在以后合并在同一OTU中)。
具体实现:
首先,嵌合体所来源的序列丰度一定要比嵌合体大。
因此,嵌合体序列是通过与丰度更高的序列进行Needleman-Wunsch 全局比对发现的,如果嵌合体的左端或者右端能与母序列毫无差别的匹配上,就认为是嵌合体。如果嵌合体序列与母序列存在单个碱基/indel的差别,也会被标记。
5.合并双端序列mergePairs()
如果两段序列确实存在overlapp,就merge起来。注意到在DADA2管道中合并是在去噪后,因此严格要求精确重叠(因为预几乎所有替代错误都已经已被降噪算法删除)。
- 注意事项
1.降噪后再合并双端序列
这是因为核心去噪算法使用了质量得分和错误率之间的经验关系。如果序列合并过后,正向,重叠和反向序列部分的这种关系会变得不一致,从而干扰降噪算法。
补充1:精确序列变异缘何替代OTU?
用精确序列变异取代OTU,这样能够使标记基因测序:
1.更加有效精确(去噪算法),
2.可重复使用(即使更多的样本进来了也不需要重算),
3.可再现且全面(不同研究之间具有可比性)。
精确序列变异有很多种叫法,都是一个意思:
Exact Sequence Variants (ESVs)
- Callahan et al. 2017 (by accident)
Amplicon Sequence Variants (ASVs)
- Needham et al. 2017
- Callahan et al. 2017
sub-OTUs (sOTUs)
- Amir et al. 2017
Zero radius OTUs (zOTUs)
- Edgar 2017
Haplotypes, oligotypes, …
其中zOTU的叫法可以形象的用下图描述:

参考
论文:dada2: high-resolution sample inference from illumina amplicon data
slides:https://www.biostat.washington.edu/sites/default/files/modules//2016-SISMID-14-01.pdf
上一篇: matplotlib绘图逻辑(上)
下一篇: 扩增子序列变异为何取代OTU